Compondo mapas em R
By Ricardo de Oliveira Perdiz in mapa R Tutorial
March 29, 2020
Esta postagem é uma reformulação de uma anterior, publicada em 2017, no blog de meu laboratório durante o doutorado (Laboratório de Ecologia e Evolução de Plantas Amazônicas, LABOTAM; veja a postagem aqui).
Para executar o tutorial abaixo e reproduzir o mapa publicado em @Rodriguesetal2017, é necessário baixar os dados presentes na página https://github.com/ricoperdiz/Tutorials/tree/master/R_map_2017_Rodriguesetal.
Baixe todos os arquivos, com exceção dos arquivos .md (markdown) e .Rmd (Rmarkdown).
Os scripts abaixo reproduzem o mapa publicado recentemente em Rodrigues, Perdiz, e Flores (2017). Caso você possua conta no ResearchGate, pode visualizar o trabalho neste link ou acessando esta página. O mapa principal apresenta localidades de coleta de espécimes de plantas em duas unidades de conservação do estado de Roraima. A maioria dessas coletas foram feitas durante a expedição Terra Incognita, ocorrida em dezembro de 2013, organizada pelas equipes gestoras do Parque Nacional Serra da Mocidade e Estação Ecológica de Niquiá, com financiamento da ARPA. Veja mais detalhes sobre a expedição ao fim desta postagem.
Carrega os pacotes
Para manipular os dados e gerar o mapa desta postagem, fiz uso dos pacotes abaixo:
- broom (Robinson, Hayes, e Couch 2022);
- dplyr (Wickham et al. 2021);
- GISTools (Brunsdon e Chen 2014);
- maps (Brownrigg 2021);
- rgdal (Bivand, Keitt, e Rowlingson 2021).
É imprescindível que todos estejam instalados, atualizados e com as dependências instaladas.
library(broom)
library(dplyr)
##
## Attaching package: 'dplyr'
## The following objects are masked from 'package:stats':
##
## filter, lag
## The following objects are masked from 'package:base':
##
## intersect, setdiff, setequal, union
library(GISTools)
## Loading required package: maptools
## Loading required package: sp
## Checking rgeos availability: TRUE
## Please note that 'maptools' will be retired by the end of 2023,
## plan transition at your earliest convenience;
## some functionality will be moved to 'sp'.
## Loading required package: RColorBrewer
## Loading required package: MASS
##
## Attaching package: 'MASS'
## The following object is masked from 'package:dplyr':
##
## select
## Loading required package: rgeos
## rgeos version: 0.5-9, (SVN revision 684)
## GEOS runtime version: 3.8.1-CAPI-1.13.3
## Please note that rgeos will be retired by the end of 2023,
## plan transition to sf functions using GEOS at your earliest convenience.
## Linking to sp version: 1.4-6
## Polygon checking: TRUE
library(maps)
##
## Attaching package: 'maps'
## The following object is masked from 'package:GISTools':
##
## map.scale
library(rgdal)
## Please note that rgdal will be retired by the end of 2023,
## plan transition to sf/stars/terra functions using GDAL and PROJ
## at your earliest convenience.
##
## rgdal: version: 1.5-28, (SVN revision 1158)
## Geospatial Data Abstraction Library extensions to R successfully loaded
## Loaded GDAL runtime: GDAL 3.2.1, released 2020/12/29
## Path to GDAL shared files: /Library/Frameworks/R.framework/Versions/4.1/Resources/library/rgdal/gdal
## GDAL binary built with GEOS: TRUE
## Loaded PROJ runtime: Rel. 7.2.1, January 1st, 2021, [PJ_VERSION: 721]
## Path to PROJ shared files: /Library/Frameworks/R.framework/Versions/4.1/Resources/library/rgdal/proj
## PROJ CDN enabled: FALSE
## Linking to sp version:1.4-6
## To mute warnings of possible GDAL/OSR exportToProj4() degradation,
## use options("rgdal_show_exportToProj4_warnings"="none") before loading sp or rgdal.
## Overwritten PROJ_LIB was /Library/Frameworks/R.framework/Versions/4.1/Resources/library/rgdal/proj
Além desses pacotes, também fiz uso dos pacotes blogdown (Xie, Dervieux, e Presmanes Hill 2022), rmarkdown (Xie, Allaire, e Grolemund 2018), e knitr (Xie 2021), tanto para gerar a estrutura deste sítio web quanto para gerar os arquivos .html que vocês estão lendo neste momento.
Importa os dados
#dados dos pontos de coleta dos vouchers
new.reg.voucher <- read.table('rodrigues_etal_2017_coordenadas_novos_registros.csv',header=T,as.is=T,sep='\t')
#kml do PARNA S Mocidade e ESEC Niquia
mocidade <- rgdal::readOGR(dsn='parna_serra_da_mocidade.kml',layer='sql_statement')
## OGR data source with driver: KML
## Source: "/Users/ricoperdiz/Documents/PROFISSIONAL/Sites/ricardoperdiz/content/blog/2020_03_29_R-composicao-mapas-Rodriguesetal2017/parna_serra_da_mocidade.kml", layer: "sql_statement"
## with 1 features
## It has 2 fields
niquia <- rgdal::readOGR(dsn='esec_niquia.kml',layer='sql_statement')
## OGR data source with driver: KML
## Source: "/Users/ricoperdiz/Documents/PROFISSIONAL/Sites/ricardoperdiz/content/blog/2020_03_29_R-composicao-mapas-Rodriguesetal2017/esec_niquia.kml", layer: "sql_statement"
## with 1 features
## It has 2 fields
#shape da Am Sul e Central
area.mapa <- rgdal::readOGR(dsn = 'SAm_CAm_shape.shp')
## OGR data source with driver: ESRI Shapefile
## Source: "/Users/ricoperdiz/Documents/PROFISSIONAL/Sites/ricardoperdiz/content/blog/2020_03_29_R-composicao-mapas-Rodriguesetal2017/SAm_CAm_shape.shp", layer: "SAm_CAm_shape"
## with 37 features
## It has 65 fields
#shape de RR
rr <- rgdal::readOGR(dsn = 'roraima.shp')
## Warning in OGRSpatialRef(dsn, layer, morphFromESRI = morphFromESRI, dumpSRS
## = dumpSRS, : Discarded datum South_American_Datum_1969 in Proj4 definition:
## +proj=longlat +ellps=aust_SA +no_defs
## OGR data source with driver: ESRI Shapefile
## Source: "/Users/ricoperdiz/Documents/PROFISSIONAL/Sites/ricardoperdiz/content/blog/2020_03_29_R-composicao-mapas-Rodriguesetal2017/roraima.shp", layer: "roraima"
## with 1 features
## It has 8 fields
## Integer64 fields read as strings: MSLINK MAPID
#shape de rios em RR
rios.main <- rgdal::readOGR(getwd(),'rios',encoding='latin1')
## OGR data source with driver: ESRI Shapefile
## Source: "/Users/ricoperdiz/Documents/PROFISSIONAL/Sites/ricardoperdiz/content/blog/2020_03_29_R-composicao-mapas-Rodriguesetal2017", layer: "rios"
## with 1849 features
## It has 16 fields
## Integer64 fields read as strings: FNODE_ TNODE_ LPOLY_ RPOLY_ RIV3M_W_ RIV3M_W_ID
rios.sec <- rgdal::readOGR(getwd(),'rios2',encoding='latin1')
## OGR data source with driver: ESRI Shapefile
## Source: "/Users/ricoperdiz/Documents/PROFISSIONAL/Sites/ricardoperdiz/content/blog/2020_03_29_R-composicao-mapas-Rodriguesetal2017", layer: "rios2"
## with 1624 features
## It has 1 fields
## Integer64 fields read as strings: FID
## Warning in rgdal::readOGR(getwd(), "rios2", encoding = "latin1"): Dropping null
## geometries: 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19,
## 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39,
## 40, 41, 42, 43, 44
Cria variáveis para os plots
# para o mapa de referencia
#amplitude de long
x1 <- c(-70,-50)
y1 <- c(-10,5)
#vetor de tamanho para o cex
cex.tam <- seq(0.8,1.6,by=0.1)
#vetor com tipos de pontos
pontos <- c(17:25)
#amplitude de lat e long - vetores
lat_long <- dplyr::full_join(tidy(mocidade), tidy(niquia))
## Regions defined for each Polygons
## Regions defined for each Polygons
## Joining, by = c("long", "lat", "order", "hole", "piece", "group", "id")
y.geral <- range(lat_long$lat) + c(-0.01,0.01)
x.geral <- range(lat_long$long) + c(-0.01,0.01)
Compõe um gráfico para acomodar os diferentes plots e plota cada mapa
À primeira vista, repare que a figura, por ser composta de três plots individuais, pode não parecer bem ajustada devido aos dispositivos de visualização:
#layout do mapa
mapa.layout <- graphics::layout(
mat = matrix(c(1,1,2,2,2,2,2,2,3,3,2,2,2,2,2,2,3,3,2,2,2,2,2,2),nrow=3,byrow=T),
widths=rep(2/8,8),
heights=rep(1/3,3),
respect=T)
# plot 1
par(mar=c(0,2,2,2))
plot(area.mapa,xlim=x1,ylim=y1,lwd=0.2)
plot(rr,add=T,col='gray90')
#plota um retangulo na area do mapa da direita
rect(xleft = min(x.geral), ybottom = min(y.geral), xright =
max(x.geral), ytop = max(y.geral), density = 25)
#coloca escala do mapa
par(cex=1, las=1)
#library(GISTools)
# inclui o NORTE
GISTools::north.arrow(max(x1)-5,max(y1)-10,len=1,cex.lab=0.9,lab='N', col = 'black')
#coloca um texto para avisar quem é Brasil e Guiana
text(-58,-10,labels='BRASIL',pos=4,cex=cex.tam[1],font=2)
box()
# plot 2
par(mar=c(2,2,2,3))
#plota o mapa das uc`s com dados específicos
plot(rr,col='gray90',xlim=x.geral,ylim=y.geral,lwd=1,lty=1) #default lwd=1,lty=1
plot(mocidade,add=T,lty=0,col='gray80')
plot(niquia,add=T,lty=0,col='gray80')
#plota os rios
plot(rios.main,add=T,col='gray60')
plot(rios.sec,add=T,col='gray60')
#plota as areas dos mapas
plot(mocidade,add=T,lwd=1.4,lty=2)
plot(niquia,add=T,lwd=1.4,lty=3)
#plota os pontos de coleta
points(new.reg.voucher$long,new.reg.voucher$lat,pch=pontos[3],col='black',cex=1.4)
#coloca nome dos rios
text(x=-61.42,y=0.8,labels='Rio Branco',pos=1,srt=60,font=3)
text(x=-61.65,y=1.3,labels='Rio Água Boa do Univini',pos=1,srt=70,font=3)
text(x=-61.97,y=0.77,labels='Rio Catrimani',pos=3,srt=-50,font=3)
text(x=-61.9,y=1.2,labels='Rio Capivara',pos=3,srt=-60,font=3)
#coloca nome das UC's
text(x=-61.80,y=1.5,labels='PARNA\nS. Mocidade',pos=1,font=2)
text(x=-61.50,y=1.3,labels='ESEC\nNiquiá',pos=1,font=2)
# coloca um texto para informar área do Amazonas e Roraima
text(-62.845,sum(range(y.geral))/2,labels='Amazonas',pos=4,cex=1.4,font=2)
text((sum(range(x.geral))/2)-0.1,max(y.geral)-0.02,labels='Roraima',pos=4,cex=1.4,font=2)
#coloca escala do mapa
par(cex=1, las=1)
maps::map.scale(max(x.geral) - 0.15, min(y.geral)+0.02, relwidth = 0.10, ratio = F, cex = 1, metric = T, col = 'black')
#coloca o Norte
GISTools::north.arrow(max(x.geral),min(y.geral)+0.07,len=0.02,lab='N',cex.lab=1.2,lwd=1.5,col='black')
#coloca eixos das coordenadas
maps::map.axes()
axis(side=4,las=1)
axis(side=3,las=1)
# plot 3 - LEGENDA
par(mar = c(1,1,0,1))
plot.new()
par(cex = 1.2)
legend(x = 'center', ncol = 1, legend =
c('Pontos de coleta dos\nvouchers','Parque Nacional Serra da\nMocidade','Estação Ecológica de\nNiquiá','Fronteira entre estados','Cursos de água'),
pch = c(pontos[3], NA, NA, NA, NA), col = c('black','black','black','black','gray60'), title = 'LEGENDA',
cex = cex.tam[3], lty = c(0,2,3,1,1), pt.lwd = 2, text.width = 0.8, lwd = 3, y.intersp = 1.8)
knitr::include_graphics("featured.png")
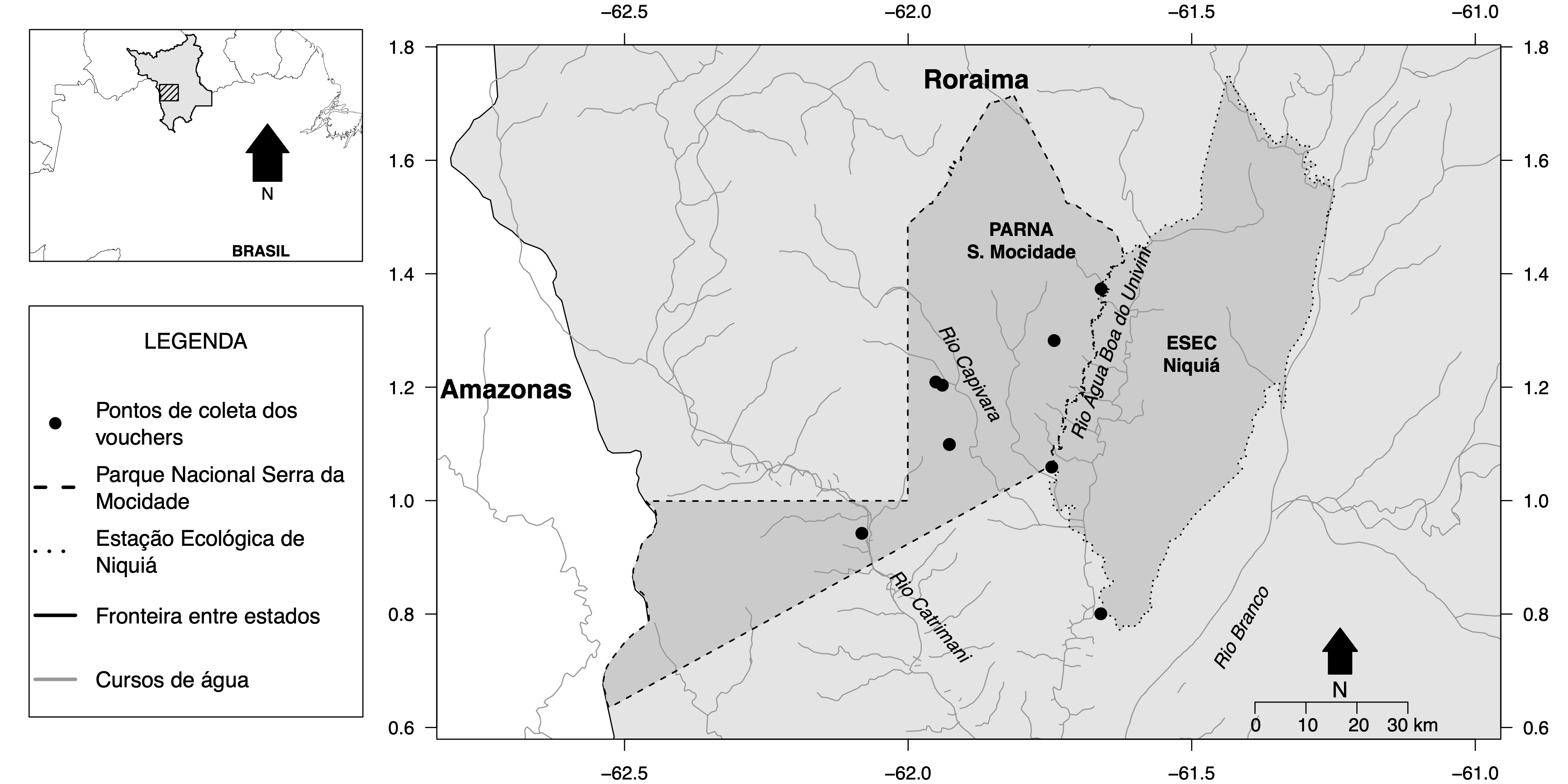
Figura 1: Mapa utilizado em Rodrigues et al. (2017) e que é o resultado deste tutorial.
Caso queira gerar um pdf com a figura acima…
Execute o código abaixo e terás, então, um pdf com a imagem acima. A mágica ocorre devido à função pdf(), inserida antes da execução da função layout.
pdf(paste('rodriguesetal2017_mapa_final.pdf',sep=''),height=7,width=14,onefile=T)
#layout do mapa
mapa.layout <- graphics::layout(
mat = matrix(c(1,1,2,2,2,2,2,2,3,3,2,2,2,2,2,2,3,3,2,2,2,2,2,2),nrow=3,byrow=T),
widths=rep(2/8,8),
heights=rep(1/3,3),
respect=T)
# plot 1
par(mar=c(0,2,2,2))
plot(area.mapa,xlim=x1,ylim=y1,lwd=0.2)
plot(rr,add=T,col='gray90')
#plota um retangulo na area do mapa da direita
rect(xleft = min(x.geral), ybottom = min(y.geral), xright =
max(x.geral), ytop = max(y.geral), density = 25)
#coloca escala do mapa
par(cex=1, las=1)
#library(GISTools)
# inclui o NORTE
GISTools::north.arrow(max(x1)-5,max(y1)-10,len=1,cex.lab=0.9,lab='N', col = 'black')
#coloca um texto para avisar quem é Brasil e Guiana
text(-58,-10,labels='BRASIL',pos=4,cex=cex.tam[1],font=2)
box()
# plot 2
par(mar=c(2,2,2,3))
#plota o mapa das uc`s com dados específicos
plot(rr,col='gray90',xlim=x.geral,ylim=y.geral,lwd=1,lty=1) #default lwd=1,lty=1
plot(mocidade,add=T,lty=0,col='gray80')
plot(niquia,add=T,lty=0,col='gray80')
#plota os rios
plot(rios.main,add=T,col='gray60')
plot(rios.sec,add=T,col='gray60')
#plota as areas dos mapas
plot(mocidade,add=T,lwd=1.4,lty=2)
plot(niquia,add=T,lwd=1.4,lty=3)
#plota os pontos de coleta
points(new.reg.voucher$long,new.reg.voucher$lat,pch=pontos[3],col='black',cex=1.4)
#coloca nome dos rios
text(x=-61.42,y=0.8,labels='Rio Branco',pos=1,srt=60,font=3)
text(x=-61.65,y=1.3,labels='Rio Água Boa do Univini',pos=1,srt=70,font=3)
text(x=-61.97,y=0.77,labels='Rio Catrimani',pos=3,srt=-50,font=3)
text(x=-61.9,y=1.2,labels='Rio Capivara',pos=3,srt=-60,font=3)
#coloca nome das UC's
text(x=-61.80,y=1.5,labels='PARNA\nS. Mocidade',pos=1,font=2)
text(x=-61.50,y=1.3,labels='ESEC\nNiquiá',pos=1,font=2)
# coloca um texto para informar área do Amazonas e Roraima
text(-62.845,sum(range(y.geral))/2,labels='Amazonas',pos=4,cex=1.4,font=2)
text((sum(range(x.geral))/2)-0.1,max(y.geral)-0.02,labels='Roraima',pos=4,cex=1.4,font=2)
#coloca escala do mapa
par(cex=1, las=1)
maps::map.scale(max(x.geral) - 0.15, min(y.geral)+0.02, relwidth = 0.10, ratio = F, cex = 1, metric = T, col = 'black')
#coloca o Norte
GISTools::north.arrow(max(x.geral),min(y.geral)+0.07,len=0.02,lab='N',cex.lab=1.2,lwd=1.5,col='black')
#coloca eixos das coordenadas
maps::map.axes()
axis(side=4,las=1)
axis(side=3,las=1)
# plot 3 - LEGENDA
par(mar = c(1,1,0,1))
plot.new()
par(cex = 1.2)
legend(x = 'center', ncol = 1, legend =
c('Pontos de coleta dos\nvouchers','Parque Nacional Serra da\nMocidade','Estação Ecológica de\nNiquiá','Fronteira entre estados','Cursos de água'),
pch = c(pontos[3], NA, NA, NA, NA), col = c('black','black','black','black','gray60'), title = 'LEGENDA',
cex = cex.tam[3], lty = c(0,2,3,1,1), pt.lwd = 2, text.width = 0.8, lwd = 3, y.intersp = 1.8)
dev.off()
Informações sobre esta postagem
Esta postagem foi escrita em ambiente R utilizando os pacotes R Markdown e knitr.
Divirta-se!!!
Sobre as unidades de conservação aqui citadas
O Parque Nacional (PARNA) Serra da Mocidade e a Estação Ecológica (ESEC) de Niquiá são unidades de conservação federal situadas no município de Caracaraí, no estado de Roraima, Brasil. O PARNA Serra da Mocidade possui uma área de 350.960 hectares e caracteriza-se pela ocorrência de uma serra isolada, com aproximadamente 1.800 metros de altitude, localizada no limite da unidade conservação com a área indígena Yanomami. Por outro lado, a ESEC Niquiá possui 284.787,42 hectares, apresentando na sua maior parte áreas planas, com duas elevações ente 200-500 m de altitude.
Sobre a expedição
No período de 04 a 18 de dezembro o Parque Nacional Serra da Mocidade e a Estação Ecológica Niquiá realizaram a Expedição de Pesquisa Científica Terra incognita, nomeada em alusão à Hamilton Rice, explorador americano que assim chamou a região oeste de Roraima em seu livro Exploração à Guiana Brasileira. Financiada com recursos do Programa ARPA (Áreas Protegidas da Amazônia), a expedição reuniu várias instituições (Instituto Chico Mendes de Conservação da Biodiversidade/ICMBio, Universidade Federal de Roraima/UFRR, Instituto de Amparo à Ciência e Tecnologia do Estado de Roraima/IACTI, Universidade Federal do Amazonas/UFAM, Centro de Estudos da Biodiversidade Amazônica/CENBAM/PPBio, Secretaria do Patrimônio da União/SPU e Instituto Federal de Roraima/IFRR) e pesquisadores de diversas áreas do conhecimento, que se propuseram a levantar informações sobre uma região pouco conhecida pela ciência, buscando contribuir com a elaboração dos planos de manejo das unidades, ora em execução.
Vejam este link para ter acesso a um pequeno documentário sobre algumas das descobertas dessa viagem.
Saiba mais
Para conferir o trabalho onde esse mapa foi publicado siga o link abaixo:
- Rodrigues et al. 2017. Novas ocorrências de angiospermas para o estado de Roraima, Brasil. Rodriguésia - Link no ResearchGate.
Para ver outros mapas produzidos da mesma maneira:
-
Lavor et al. 2016. Rediscovery of Pilosocereus oligolepsis (Cactaceae) in the State of Roraima, Brazil. Cactus and Succulent Journal 88(3):137-143 - Link no ResearchGate;
-
Barbosa et al. 2017. Decomposition rates of coarse woody debris in undisturbed Amazonian seasonally flooded and unflooded forests in the Rio Negro-Rio Branco Basin in Roraima, Brazil. Forest Ecology and Management 397(1) - Link no ResearchGate.
Referências
Bivand, Roger, Tim Keitt, e Barry Rowlingson. 2021. rgdal: Bindings for the Geospatial Data Abstraction Library. https://CRAN.R-project.org/package=rgdal.
Brownrigg, Ray. 2021. maps: Draw Geographical Maps. https://CRAN.R-project.org/package=maps.
Brunsdon, Chris, e Hongyan Chen. 2014. GISTools: Some further GIS capabilities for R. https://CRAN.R-project.org/package=GISTools.
Robinson, David, Alex Hayes, e Simon Couch. 2022. broom: Convert Statistical Objects into Tidy Tibbles. https://CRAN.R-project.org/package=broom.
Rodrigues, Rodrigo Schütz, Ricardo Oliveira Perdiz, e Andréia Silva Flores. 2017. “Novas ocorrências de angiospermas para o estado de Roraima, Brasil”. Rodriguésia 68 (2): 783–790. https://doi.org/10.1590/2175-7860201768229.
Wickham, Hadley, Romain François, Lionel Henry, e Kirill Müller. 2021. dplyr: A Grammar of Data Manipulation. https://CRAN.R-project.org/package=dplyr.
Xie, Yihui. 2021. knitr: A General-Purpose Package for Dynamic Report Generation in R. https://yihui.org/knitr/.
Xie, Yihui, J. J. Allaire, e Garrett Grolemund. 2018. R Markdown: The Definitive Guide. Boca Raton, Florida: Chapman; Hall/CRC. https://bookdown.org/yihui/rmarkdown.
Xie, Yihui, Christophe Dervieux, e Alison Presmanes Hill. 2022. blogdown: Create Blogs and Websites with R Markdown.
- Posted on:
- March 29, 2020
- Length:
- 10 minute read, 2034 words