Visualização de árvores filogenéticas
Com {ggtree}
By Ricardo Perdiz in rmarkdown R
February 28, 2022
Objetivo
Demonstrarei como utilizar os pacotes treeio (Wang et al. 2020; Yu 2023b), tidytree (Yu 2023a) e ggtree (Yu et al. 2017; Yu, Lam, e Xu 2023) para importar árvores filogenéticas, manipulá-las e visualizá-las dentro do R, respectivamente. Existem alternativas consagradas na linguagem para executar a importação, manipulação e visualização de árvores filogenéticas (ape: Paradis e Schliep (2019) e Paradis et al. (2023); phytools: Revell (2012) e Revell (2023)), porém, como tenho utilizado o ggtree há algum tempo, eu decidi demonstrar algumas de suas funcionalidades e deixar um registro tanto para mim quanto para os interessados.
Esta postagem se concentrará apenas na parte de visualização. Pretendo fazer outras cobrindo os tópicos de importação de dados filogenéticos e alinhamentos, e anotações em árvores filogenéticas.
Lembro aos leitores que isto não é um curso de R, portanto, caso tenha alguma dúvida, deixe um comentário ou busque aprender mais a manipular a linguagem em nosso curso de R, disponível gratuitamente em https://intror.netlify.app. Temos também uma lista de vídeos ensinando passo a passo algumas das aulas deste curso. Acessem essa lista de vídeos neste endereço: https://youtube.com/playlist?list=PL4WeYnT3-QHNrCuwsifcvKoivXQdfCPZ4.
É imprescindível que os pacotes utilizados nesta postagem estejam instalados, atualizados e com as dependências instaladas. Além disso, não custa lembrar também de que você deve ter o R instalado em seu computador (versão mínima 4.0).
Sugiro a leitura do livro Data Integration, Manipulation and Visualization of Phylogenetic Trees de autoria do professor Dr. Guangchuang Yu, autor do pacote ggtree, objeto desta postagem. O livro é extremamente detalhado e possui uma extensa série de exemplos no capítulo 4, o qual serve de modelo para esta postagem.
Antes de iniciar, algumas dicas

Jovem padawan, exerça sua calma e paciência.
Trabalhar com o R não é fácil caso você não se dedique diariamente, exige muita dedicação para que você se sinta à vontade. Busquem aprender a lidar com a frustração dos recorrentes erros que aparecem. Leiam a seção 1.7 de nosso livro “ Curso introdutório ao R” que apresenta uma lista de erros comuns de sintaxe da linguagem R para checar se o que apareceu para vocês se enquadra em alguma das explicações.
Quando forem lidar com a geração de arquivos .pdf, .jpeg e afins, é importante que os arquivos sejam salvos com diferentes configurações de tamanho pois é somente com o arquivo pronto que você será capaz de ver se a imagem ficou boa, se os nomes estão legíveis, se a espessura dos ramos facilita a visualização, e assim por diante.
Então, fica a dica, jovem padawan: não desistam, tentem e tentem e tentem, sem parar. E testem, testem, testem sem parar ✊ ✊ ✊. Em caso de dúvidas, escrevam ✍️, ✍.
Que a Forca esteja com você, jovem padawan!

Instalando os pacotes ggtree, tidytree e treeio
Os pacotes ggtree, tidytree e treeio não estão disponíveis no CRAN, logo, é necessário instalá-los utilizando o BioConductor, um repositório de pacotes R voltados para a análise de dados biológicos.
É necessário primeiro instalar o pacote BiocManager (Morgan e Ramos 2023) direto do CRAN usando a função install.packages().
O comando abaixo checa se o pacote BiocManager existe em seu computador.
Caso não esteja, o comando install.packages("BiocManager") instalará o pacote.
if (!require("BiocManager", quietly = TRUE)) {
install.packages("BiocManager")
}
Em seguida, execute o comando abaixo para instalar os pacotes ggtree, tidytree e treeio:
BiocManager::install(c("ggtree", "tidytree","treeio"))
Eu recomendo também que sejam instalados os pacotes ggplot2 (Wickham 2016; Wickham et al. 2023) e cowplot (Wilke 2020).
install.packages(c("cowplot", "ggplot2"))
Carregando os pacotes
Depois de ter os pacotes citados acima instalados, vamos carregá-los à sessão de trabalho utilizando a função library().
library("ggtree")
library("tidytree")
library("treeio")
library("ggplot2")
library("cowplot")
Dados para nossa prática
Vamos utilizar os dados de Paul V. A. Fine, Zapata, e Daly (2014), baixados do repositório
Dryad no seguinte endereço:
https://datadryad.org/stash/dataset/doi:10.5061/dryad.d71c1 (Paul Van Antwerp Fine, Zapata, e Daly 2014).
Este conjunto de dados contem uma árvore filogenética em formato .nex.
Para ler esta árvore, vamos utilizar a função read.beast() do pacote treeio:
arvore <- treeio::read.beast("S15484.nex")
Após ler o arquivo, nós atribuímos o resultado da leitura ao objeto arvore.
Vamos ver os dados dessa árvore:
arvore
## 'treedata' S4 object that stored information of
## 'S15484.nex'.
##
## ...@ phylo:
##
## Phylogenetic tree with 118 tips and 117 internal nodes.
##
## Tip labels:
## Protium_javanicum, Protium_pecuniosum, Protium_costaricense,
## Protium_confusum, Protium_sessiliflorum, Protium_panamense, ...
##
## Rooted; includes branch lengths.
Dados lidos com as funções do pacote treeio retornam objetos de classe treedata, que nada mais são que
objetos de classe S4, um tipo especial de sistema de objetos dentro da linguagem R.
Em resumo, esse objeto oferece uma agregação de dados em torno de um único objeto.
Reparem que a mensagem acima aponta a informação ...@ phylo:.
Se nós executarmos o comando arvore@phylo, vejamos o que acontece:
arvore@phylo
##
## Phylogenetic tree with 118 tips and 117 internal nodes.
##
## Tip labels:
## Protium_javanicum, Protium_pecuniosum, Protium_costaricense, Protium_confusum, Protium_sessiliflorum, Protium_panamense, ...
##
## Rooted; includes branch lengths.
Apenas os dados referentes à árvore são retornados. Nela, vemos que a árvore está enraizada, possui tamanho dos ramos, e existem 118 terminais.
Por trabalharmos com objetos criados dentro de uma mesma filosofia, não precisaremos utilizar a notação arvore@phylo para lidar com os dados filogenéticos.
As funções do pacote lidarão automaticamente com objetos de classe treedata.
Checando os dados
Antes de prosseguirmos com a visualização da árvore, vamos checar algumas coisas possíveis de serem feitas.
Nomes dos terminais
arvore@phylo$tip.label
## [1] "Protium_javanicum" "Protium_pecuniosum"
## [3] "Protium_costaricense" "Protium_confusum"
## [5] "Protium_sessiliflorum" "Protium_panamense"
## [7] "Tetragastris_panamensis" "Protium_obtusifolium"
## [9] "Protium_altsonii" "Protium_elegans"
## [11] "Protium_glabrescens" "Protium_heptaphyllum_ulei"
## [13] "Protium_heptaphyllum_1" "Protium_sagotianum"
## [15] "Protium_ravenii" "Protium_demerarense"
## [17] "Protium_cuneatum" "Protium_spruceanum"
## [19] "Protium_crassipetalum" "Protium_macgregori"
## [21] "Protium_glaucescens" "Protium_pallidum"
## [23] "Protium_reticulatum" "Protium_carolense"
## [25] "Protium_sp_nov_aff_opacum" "Protium_aracouchini"
## [27] "Protium_alvarezianum" "Protium_subserratum_MPH1"
## [29] "Protium_subserratum_MPH2" "Protium_subserratum_MPH3"
## [31] "Protium_gallosum" "Protium_laxiflorum"
## [33] "Protium_divaricatum_divaricatum" "Protium_nodulosum"
## [35] "Protium_paniculatum_paniculatum" "Protium_opacum"
## [37] "Protium_klugii" "Protium_aidanianum"
## [39] "Protium_copal" "Crepidospermum_goudotianum"
## [41] "Protium_ferrugineum" "Crepidospermum_prancei"
## [43] "Protium_divaricatum_fumarium" "Protium_plagiocarpium"
## [45] "Protium_trifoliolatum" "Protium_gallicum"
## [47] "Protium_sp_nov_aff_apiculatum" "Protium_guianense"
## [49] "Protium_sp_nov_G1" "Protium_decandrum"
## [51] "Protium_giganteum" "Protium_unifoliatum"
## [53] "Protium_calanense" "Protium_urophyllidium"
## [55] "Protium_strumosum" "Protium_attenuatum"
## [57] "Protium_madagascariense" "Protium_carnosum"
## [59] "Protium_rhyncophyllum" "Protium_sp_nov_Aff_meridionale"
## [61] "Protium_rubrum" "Tetragastris_altissima"
## [63] "Protium_sp_nov_Pich" "Protium_puncticulatum"
## [65] "Protium_leptostachyum" "Crepidospermum_sp_nov"
## [67] "Protium_paniculatum_riedelianum" "Protium_paniculatum_modestum"
## [69] "Protium_nitidifolium" "Protium_heptaphyllum_heptaphyllum"
## [71] "Protium_grandifolium" "Protium_polybotryum"
## [73] "Protium_polybotryum_blackii" "Protium_pittieri"
## [75] "Crepidospermum_atlanticum" "Tetragastris_breviacuminata"
## [77] "Protium_icicariba" "Protium_heptaphyllum_2"
## [79] "Protium_glabrum" "Protium_brasiliense"
## [81] "Protium_warmingianum" "Protium_glaziovii"
## [83] "Protium_kleinii" "Protium_ovatum"
## [85] "Tetragastris_balsamifera" "Protium_serratum"
## [87] "Protium_sp_nov_G5" "Tetragastris_hostmannii"
## [89] "Tetragastris_catuaba" "Protium_widgrenii"
## [91] "Protium_hebetatum" "Protium_subacuminatum"
## [93] "Protium_maestrense" "Protium_fragrans"
## [95] "Protium_baracoense" "Protium_cubense"
## [97] "Protium_krukovii" "Protium_amazonicum"
## [99] "Protium_divaricatum_krukoffii" "Protium_apiculatum"
## [101] "Protium_morii" "Protium_glomerulosum"
## [103] "Protium_amplum" "Protium_nervosum"
## [105] "Crepidospermum_rhoifolium" "Protium_colombianum"
## [107] "Protium_sp_nov_Pap" "Tetragastris_varians"
## [109] "Protium_cranipyrenum" "Protium_pilosum"
## [111] "Protium_calendulinum" "Boswellia_sacra"
## [113] "Bursera_simaruba" "Canarium_pilosum"
## [115] "Commiphora_edulis" "Pistacia_mexicana"
## [117] "Rhus_trilobata" "Santiria_griffithii"
Como tudo é um objeto no R, podemos manipular esses nomes.
Por exemplo, podemos retirar o underline dos nomes.
Façamos assim:
gsub("_", " ", arvore@phylo$tip.label)
## [1] "Protium javanicum" "Protium pecuniosum"
## [3] "Protium costaricense" "Protium confusum"
## [5] "Protium sessiliflorum" "Protium panamense"
## [7] "Tetragastris panamensis" "Protium obtusifolium"
## [9] "Protium altsonii" "Protium elegans"
## [11] "Protium glabrescens" "Protium heptaphyllum ulei"
## [13] "Protium heptaphyllum 1" "Protium sagotianum"
## [15] "Protium ravenii" "Protium demerarense"
## [17] "Protium cuneatum" "Protium spruceanum"
## [19] "Protium crassipetalum" "Protium macgregori"
## [21] "Protium glaucescens" "Protium pallidum"
## [23] "Protium reticulatum" "Protium carolense"
## [25] "Protium sp nov aff opacum" "Protium aracouchini"
## [27] "Protium alvarezianum" "Protium subserratum MPH1"
## [29] "Protium subserratum MPH2" "Protium subserratum MPH3"
## [31] "Protium gallosum" "Protium laxiflorum"
## [33] "Protium divaricatum divaricatum" "Protium nodulosum"
## [35] "Protium paniculatum paniculatum" "Protium opacum"
## [37] "Protium klugii" "Protium aidanianum"
## [39] "Protium copal" "Crepidospermum goudotianum"
## [41] "Protium ferrugineum" "Crepidospermum prancei"
## [43] "Protium divaricatum fumarium" "Protium plagiocarpium"
## [45] "Protium trifoliolatum" "Protium gallicum"
## [47] "Protium sp nov aff apiculatum" "Protium guianense"
## [49] "Protium sp nov G1" "Protium decandrum"
## [51] "Protium giganteum" "Protium unifoliatum"
## [53] "Protium calanense" "Protium urophyllidium"
## [55] "Protium strumosum" "Protium attenuatum"
## [57] "Protium madagascariense" "Protium carnosum"
## [59] "Protium rhyncophyllum" "Protium sp nov Aff meridionale"
## [61] "Protium rubrum" "Tetragastris altissima"
## [63] "Protium sp nov Pich" "Protium puncticulatum"
## [65] "Protium leptostachyum" "Crepidospermum sp nov"
## [67] "Protium paniculatum riedelianum" "Protium paniculatum modestum"
## [69] "Protium nitidifolium" "Protium heptaphyllum heptaphyllum"
## [71] "Protium grandifolium" "Protium polybotryum"
## [73] "Protium polybotryum blackii" "Protium pittieri"
## [75] "Crepidospermum atlanticum" "Tetragastris breviacuminata"
## [77] "Protium icicariba" "Protium heptaphyllum 2"
## [79] "Protium glabrum" "Protium brasiliense"
## [81] "Protium warmingianum" "Protium glaziovii"
## [83] "Protium kleinii" "Protium ovatum"
## [85] "Tetragastris balsamifera" "Protium serratum"
## [87] "Protium sp nov G5" "Tetragastris hostmannii"
## [89] "Tetragastris catuaba" "Protium widgrenii"
## [91] "Protium hebetatum" "Protium subacuminatum"
## [93] "Protium maestrense" "Protium fragrans"
## [95] "Protium baracoense" "Protium cubense"
## [97] "Protium krukovii" "Protium amazonicum"
## [99] "Protium divaricatum krukoffii" "Protium apiculatum"
## [101] "Protium morii" "Protium glomerulosum"
## [103] "Protium amplum" "Protium nervosum"
## [105] "Crepidospermum rhoifolium" "Protium colombianum"
## [107] "Protium sp nov Pap" "Tetragastris varians"
## [109] "Protium cranipyrenum" "Protium pilosum"
## [111] "Protium calendulinum" "Boswellia sacra"
## [113] "Bursera simaruba" "Canarium pilosum"
## [115] "Commiphora edulis" "Pistacia mexicana"
## [117] "Rhus trilobata" "Santiria griffithii"
Podemos atribuir essa mudança aos dados originais.
Caso façamos isso, os nomes dos terminais a partir de agora serão vistos sem o _.
Façamos:
arvore@phylo$tip.label <- gsub("_", " ", arvore@phylo$tip.label)
Para ver se deu certo, vamos executar novamente o objeto arvore e ver o resultado.
Comparem o resultado desta ação com o que executamos anteriormente.
Vejam como os nomes das espécies agora não apresentam o _.
arvore
## 'treedata' S4 object that stored information of
## 'S15484.nex'.
##
## ...@ phylo:
##
## Phylogenetic tree with 118 tips and 117 internal nodes.
##
## Tip labels:
## Protium javanicum, Protium pecuniosum, Protium costaricense, Protium
## confusum, Protium sessiliflorum, Protium panamense, ...
##
## Rooted; includes branch lengths.
Número de nós internos
arvore@phylo$Nnode
## [1] 117
Plotando a árvore
O primeiro passo para plotar uma árvore filogenética é usar o nosso objeto arvore dentro da função ggtree().
ggtree(arvore)
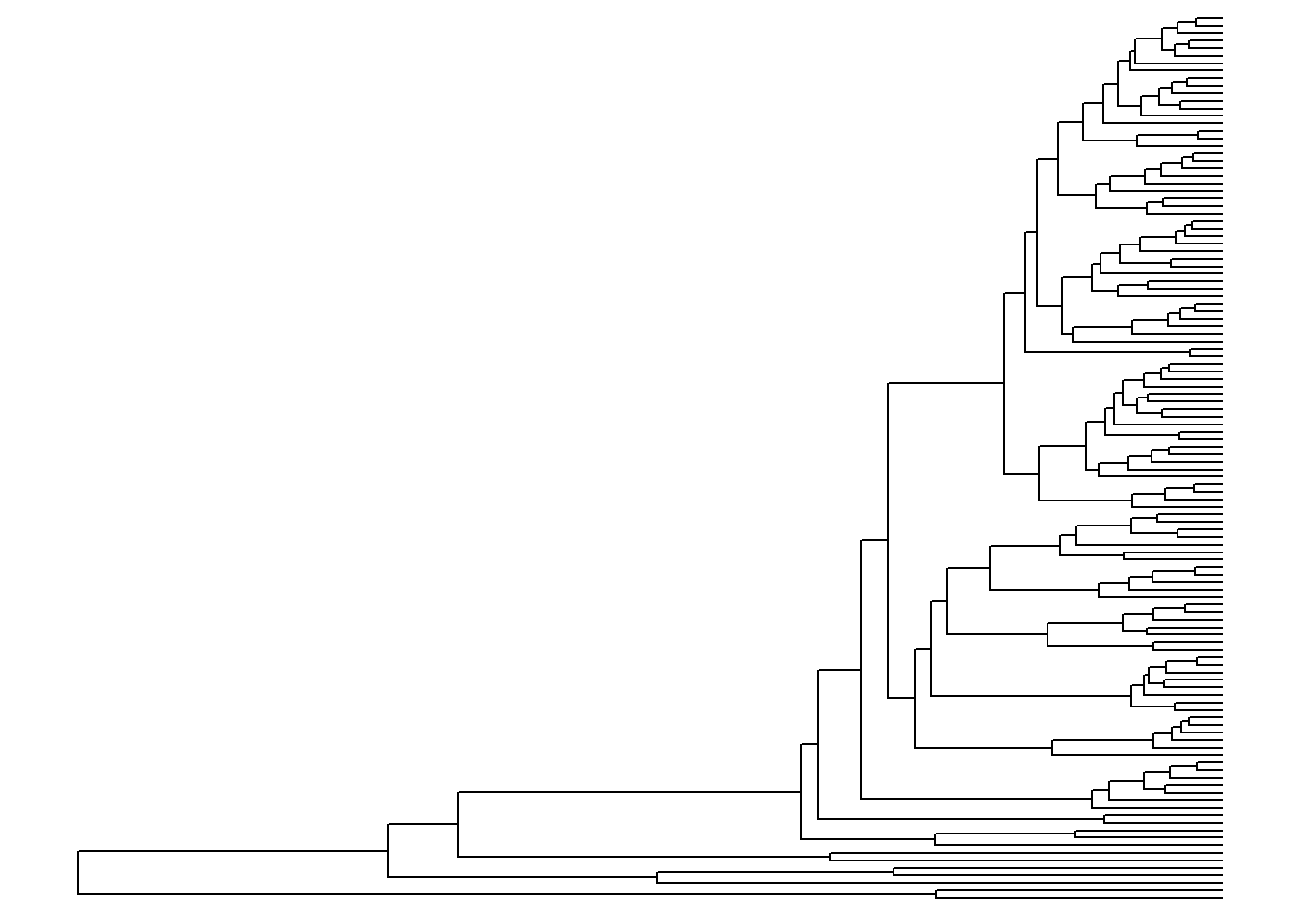
Imediatamente, é plotada uma árvore. Por padrão, ela aparece em forma de escada (ladderized em inglês).
Podemos atribuir esta árvore a um objeto de nome p.
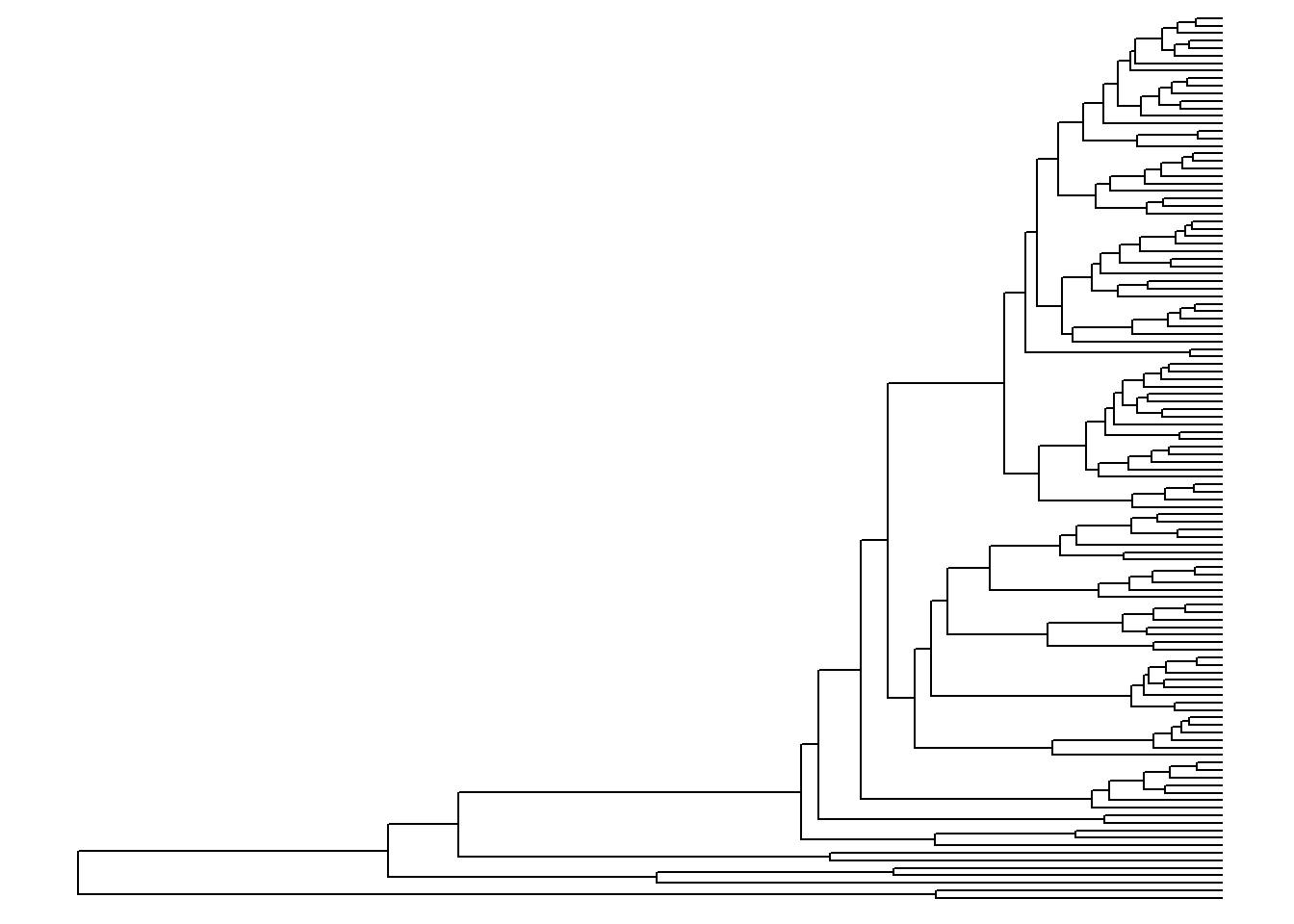
Ao fazer isto, teremos que p sempre conterá o resultado de ggtree(arvore).
p <- ggtree(arvore)
p
Formas da árvore
Podemos visualizar a árvore de diversas maneiras, utilizando as diversas opções do argumento layout.
Leiam o ? da função para entender seus argumentos, possibilidades, e usos.
Executem ?ggtree no console e tenham acesso à descrição da função, argumentos, e usos.
Vejam abaixo algumas das possíveis visualizações:
ggtree(arvore, ladderize = FALSE)
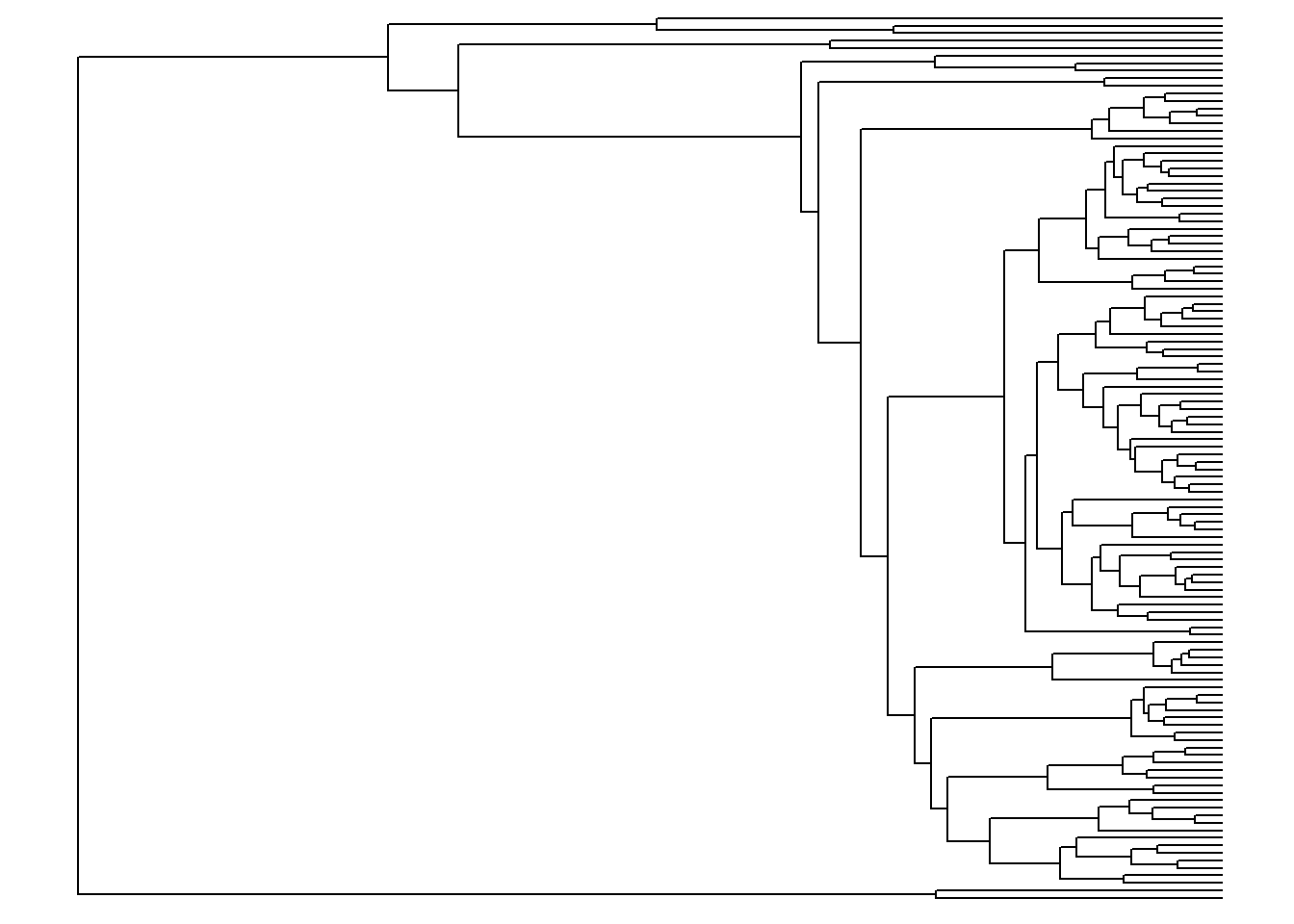
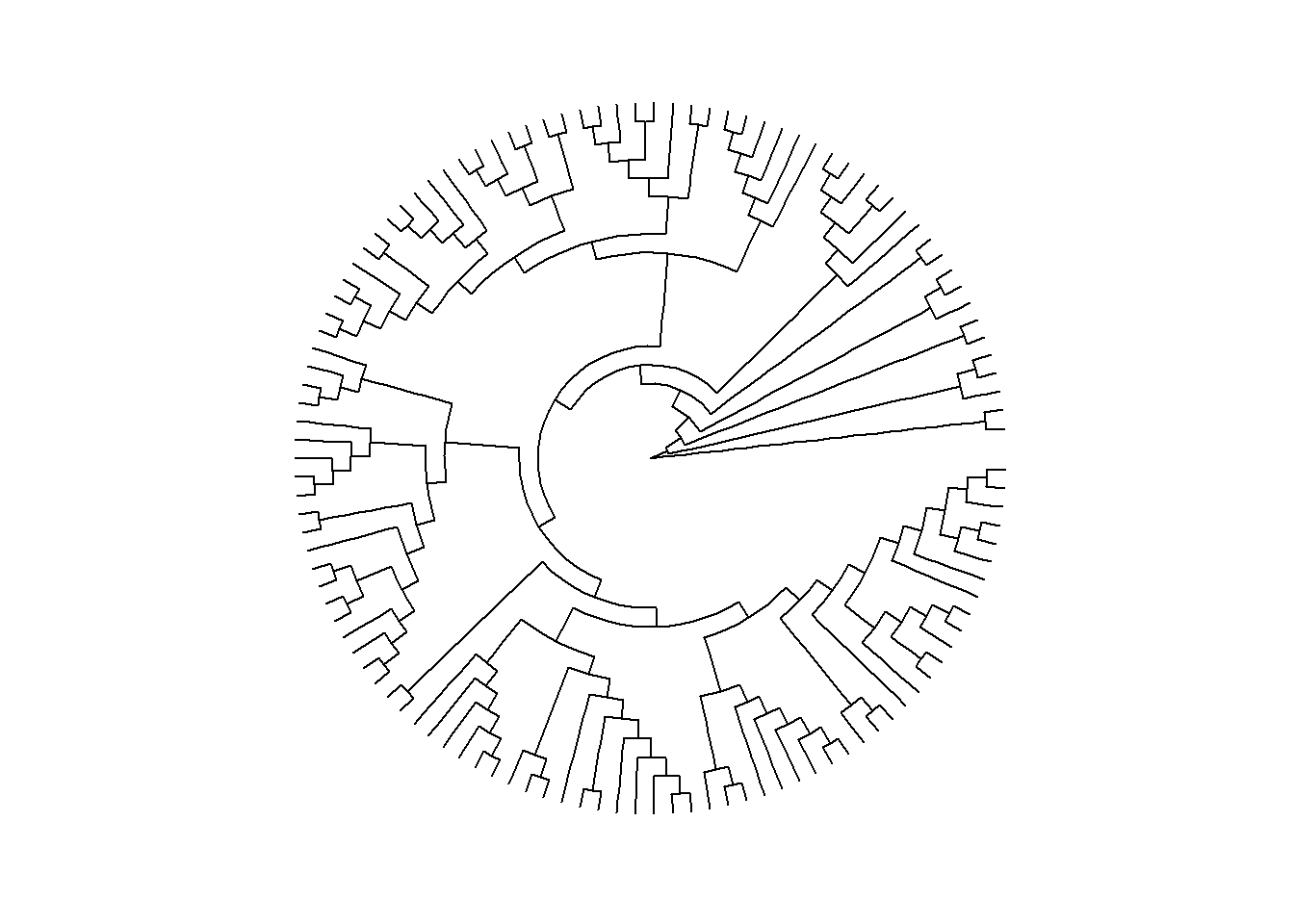
ggtree(arvore, layout = "circular")

ggtree(arvore, layout = "roundrect")
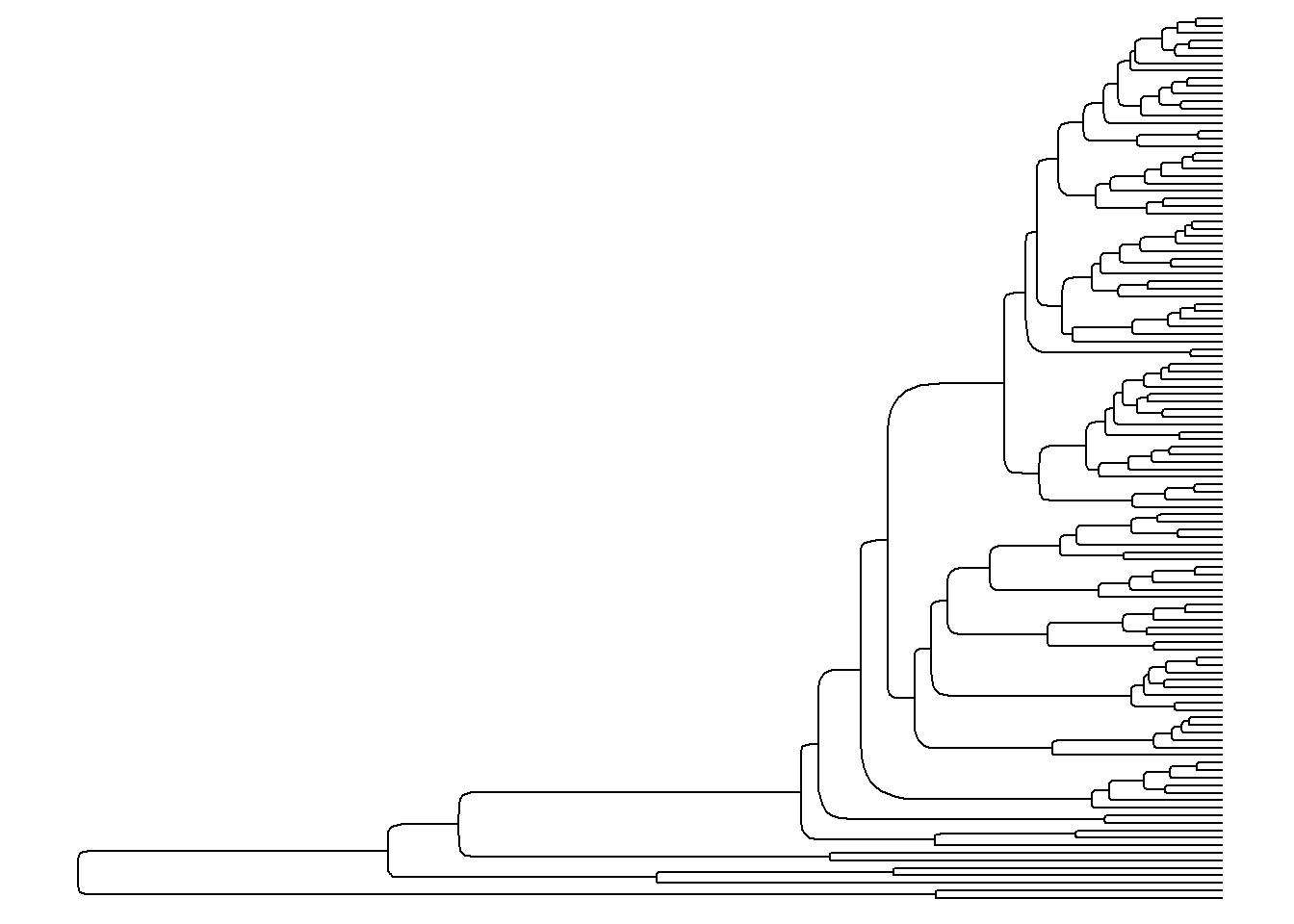
ggtree(arvore, layout = "slanted")
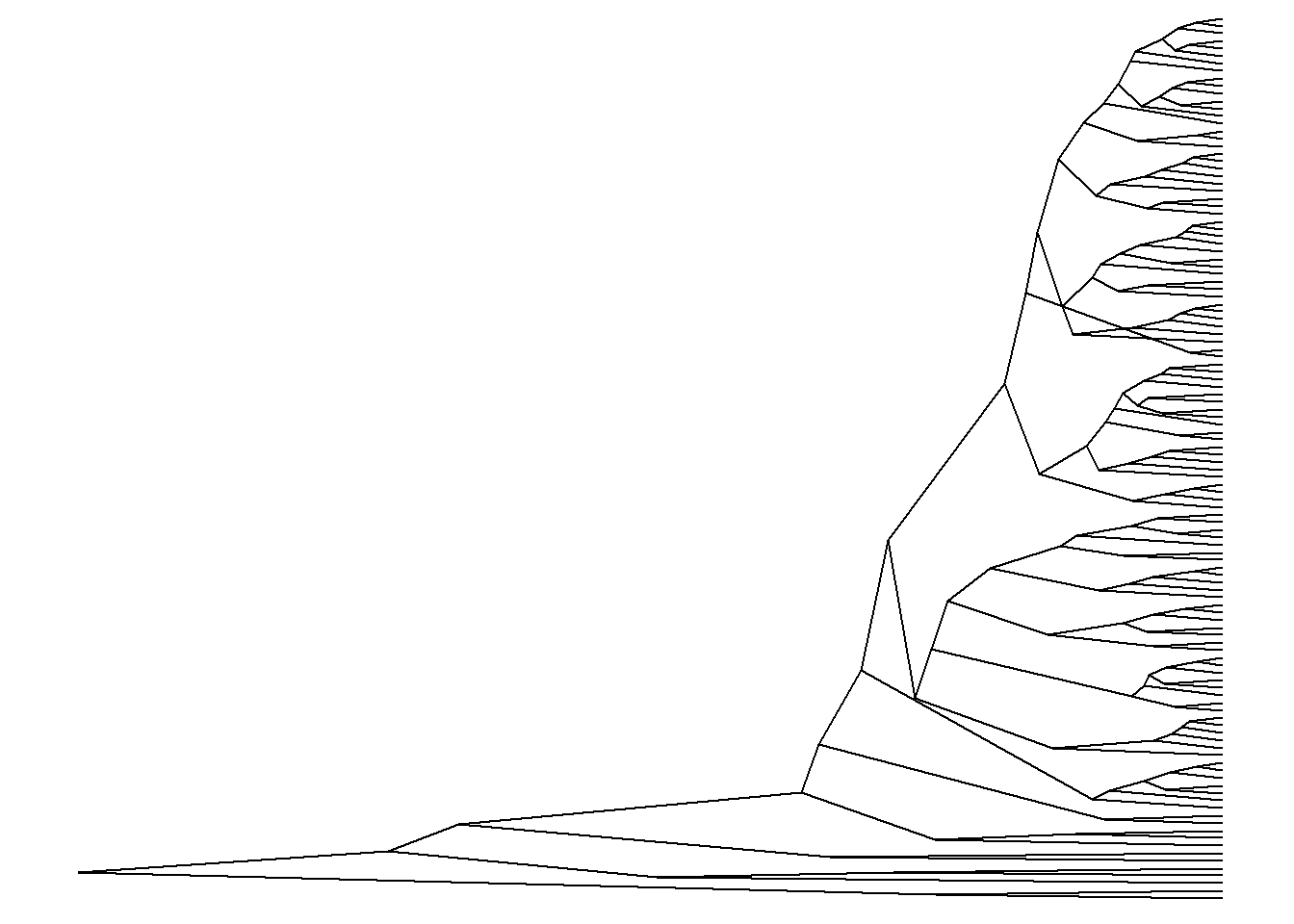
ggtree(arvore, layout = "ellipse")

ggtree(arvore, layout = "fan", open.angle = 120)
## Scale for y is already present.
## Adding another scale for y, which will replace the existing scale.
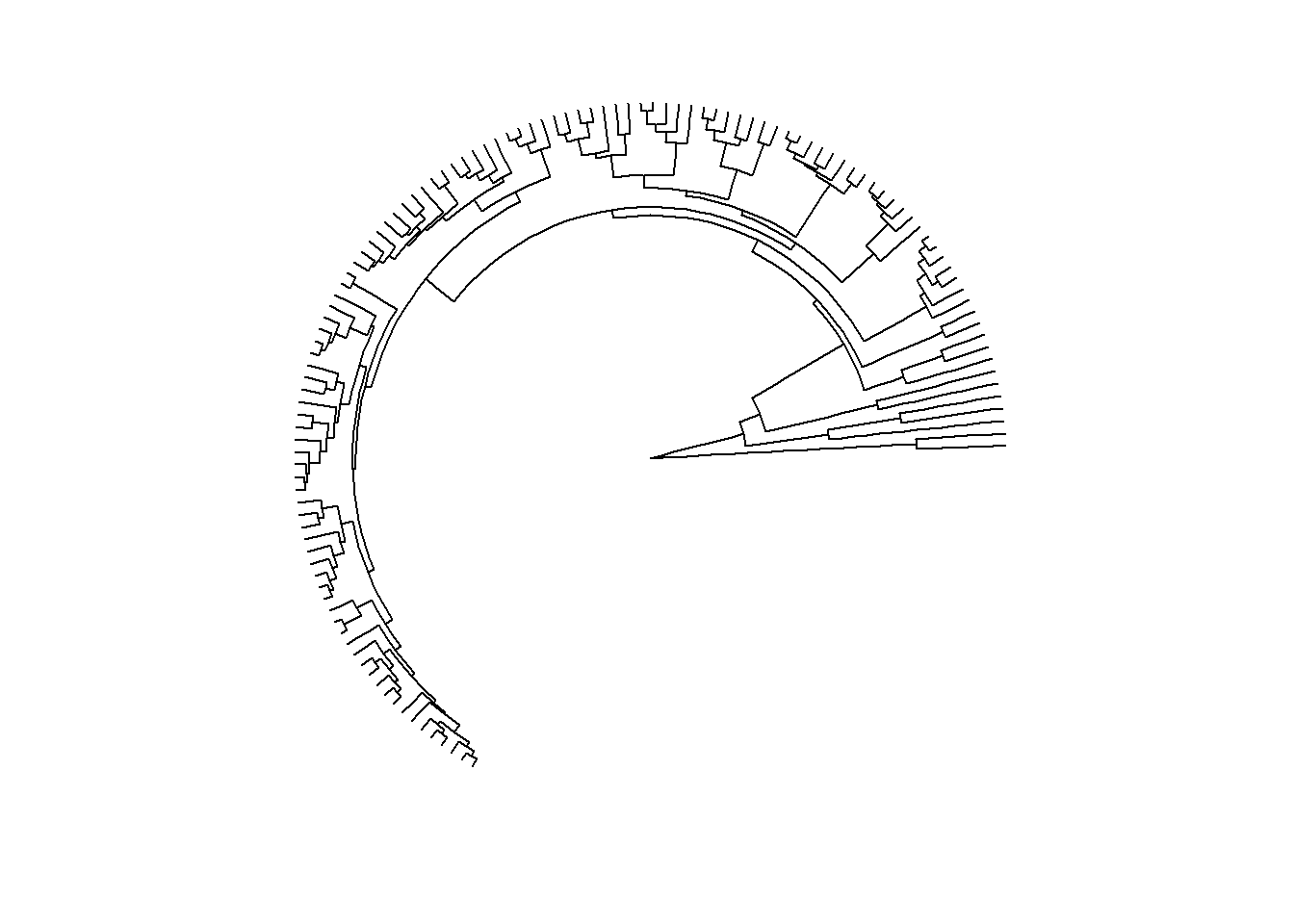
ggtree(arvore, layout = "equal_angle")
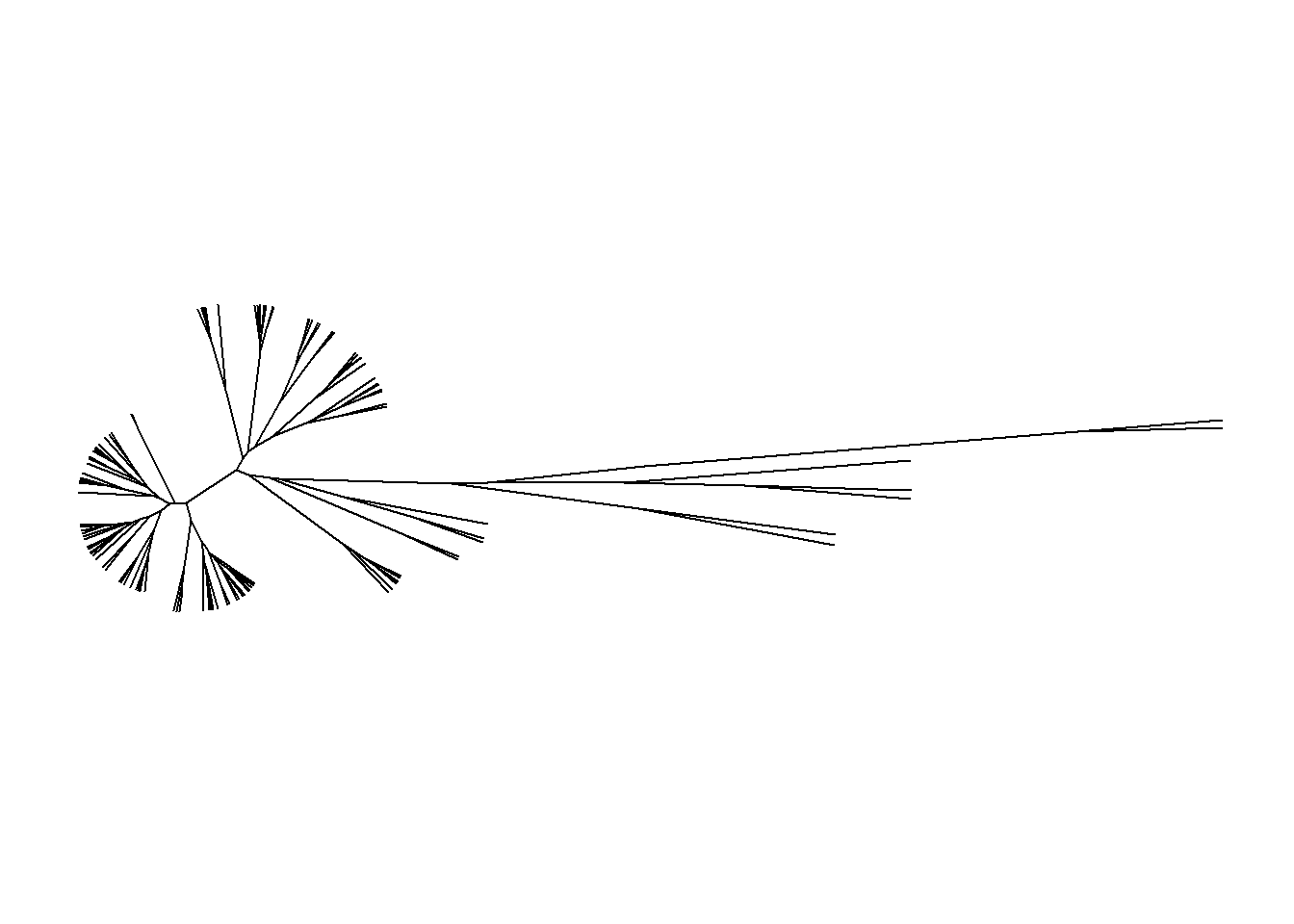
ggtree(arvore, layout = "daylight")
## Average angle change [1] 0.0672566652386173
## Average angle change [2] 0.0468278266057616
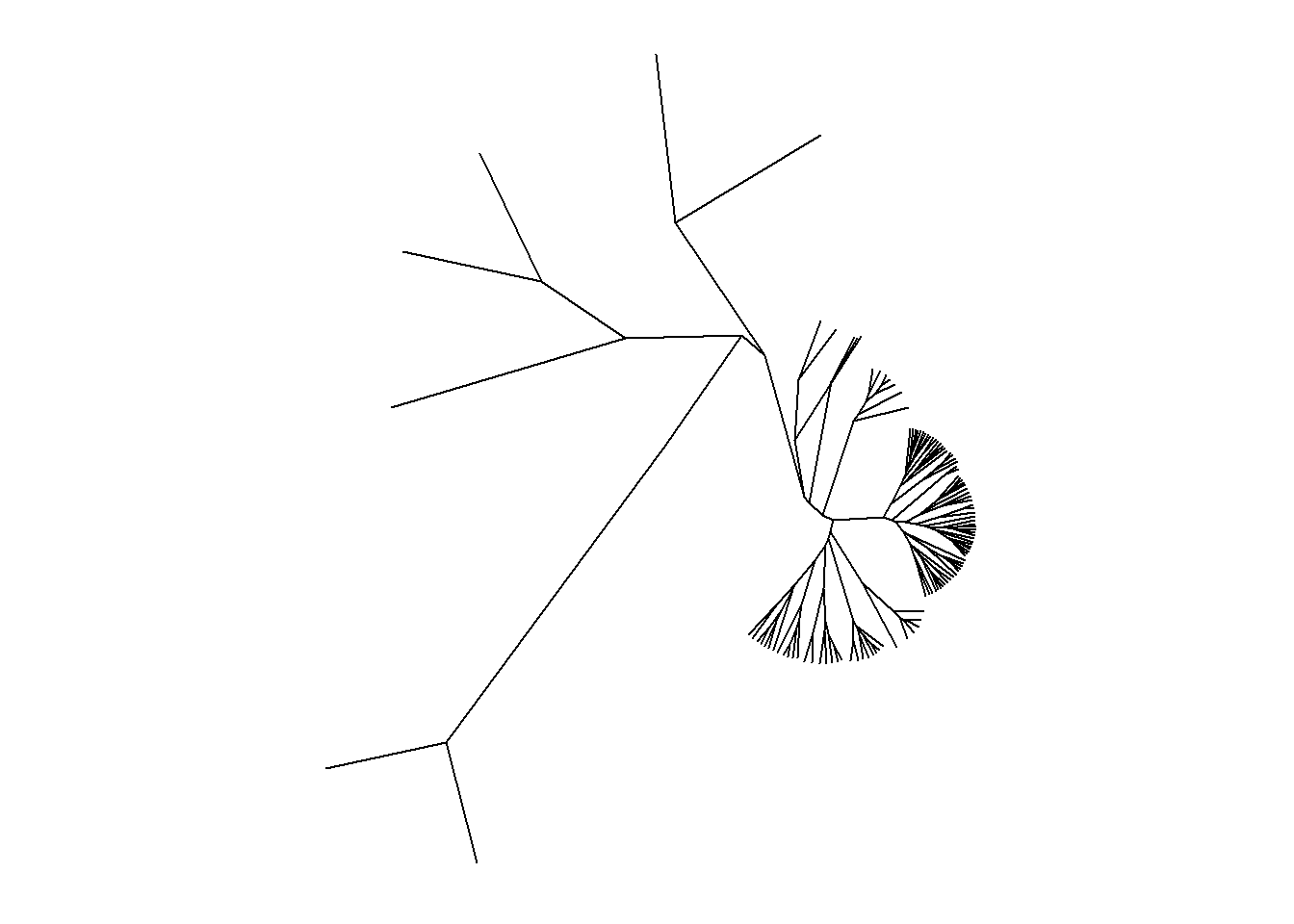
ggtree(arvore, branch.length = "none")
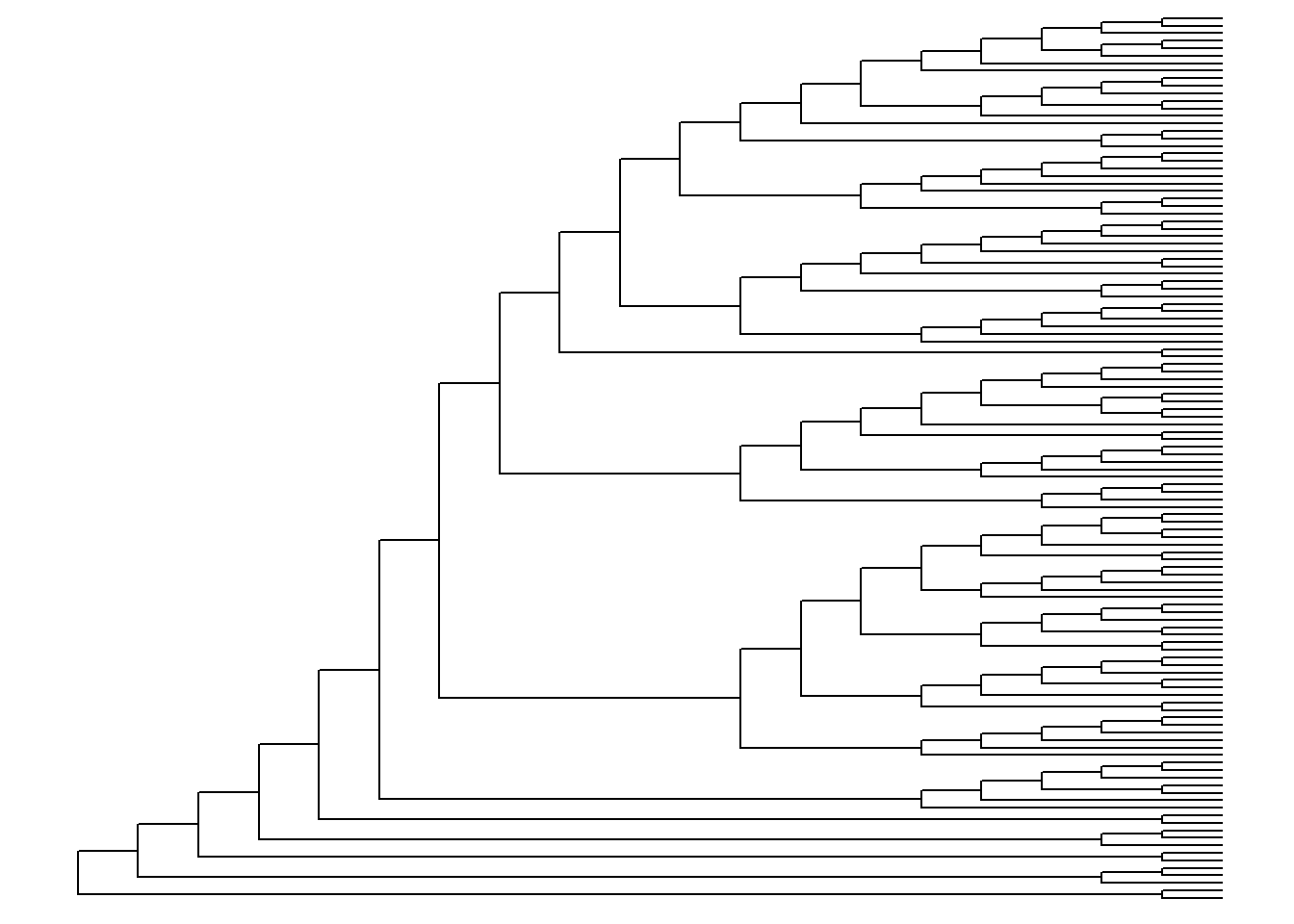
ggtree(arvore, layout = "ellipse", branch.length = "none")
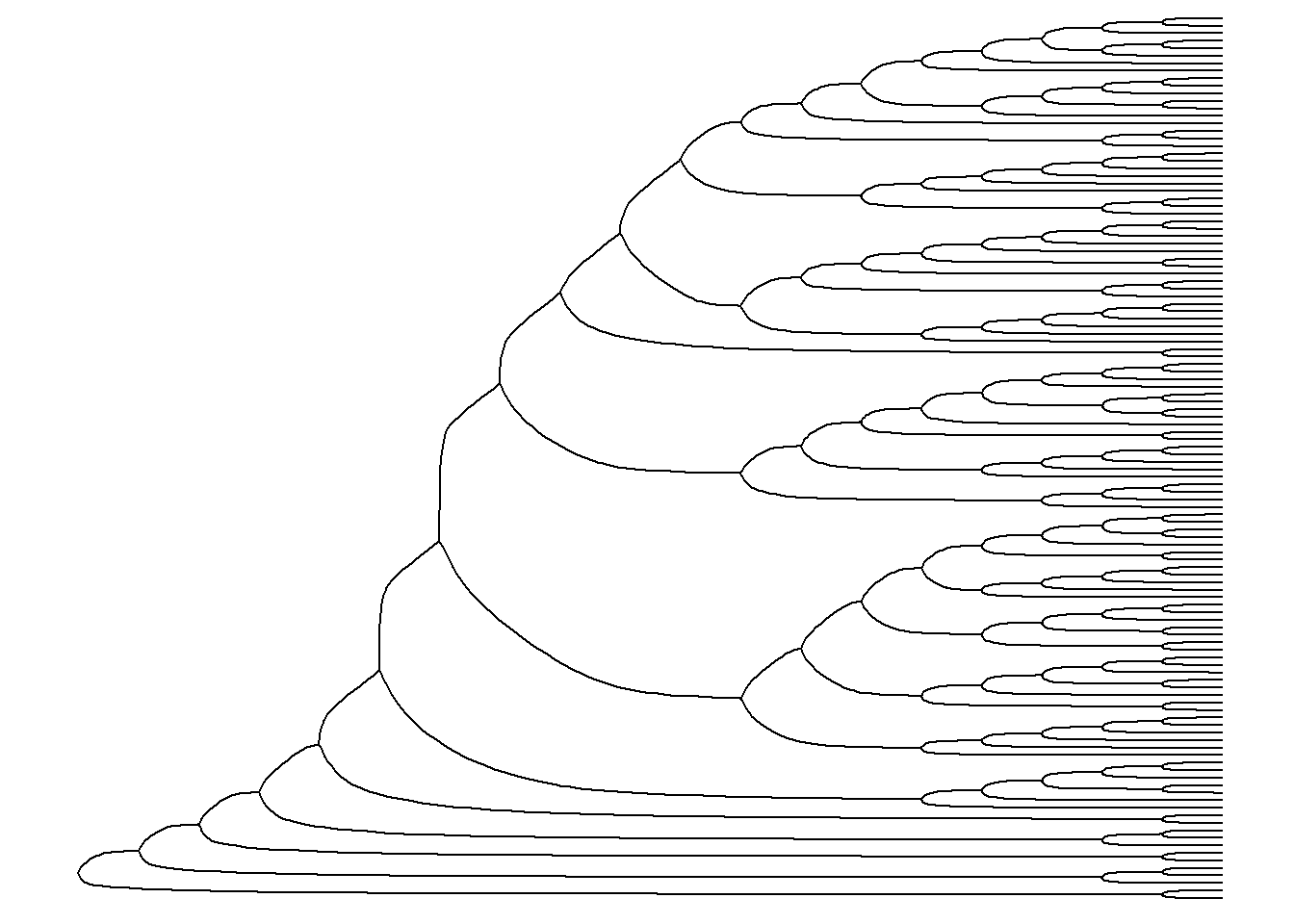
ggtree(arvore, branch.length = "none", layout = "circular")
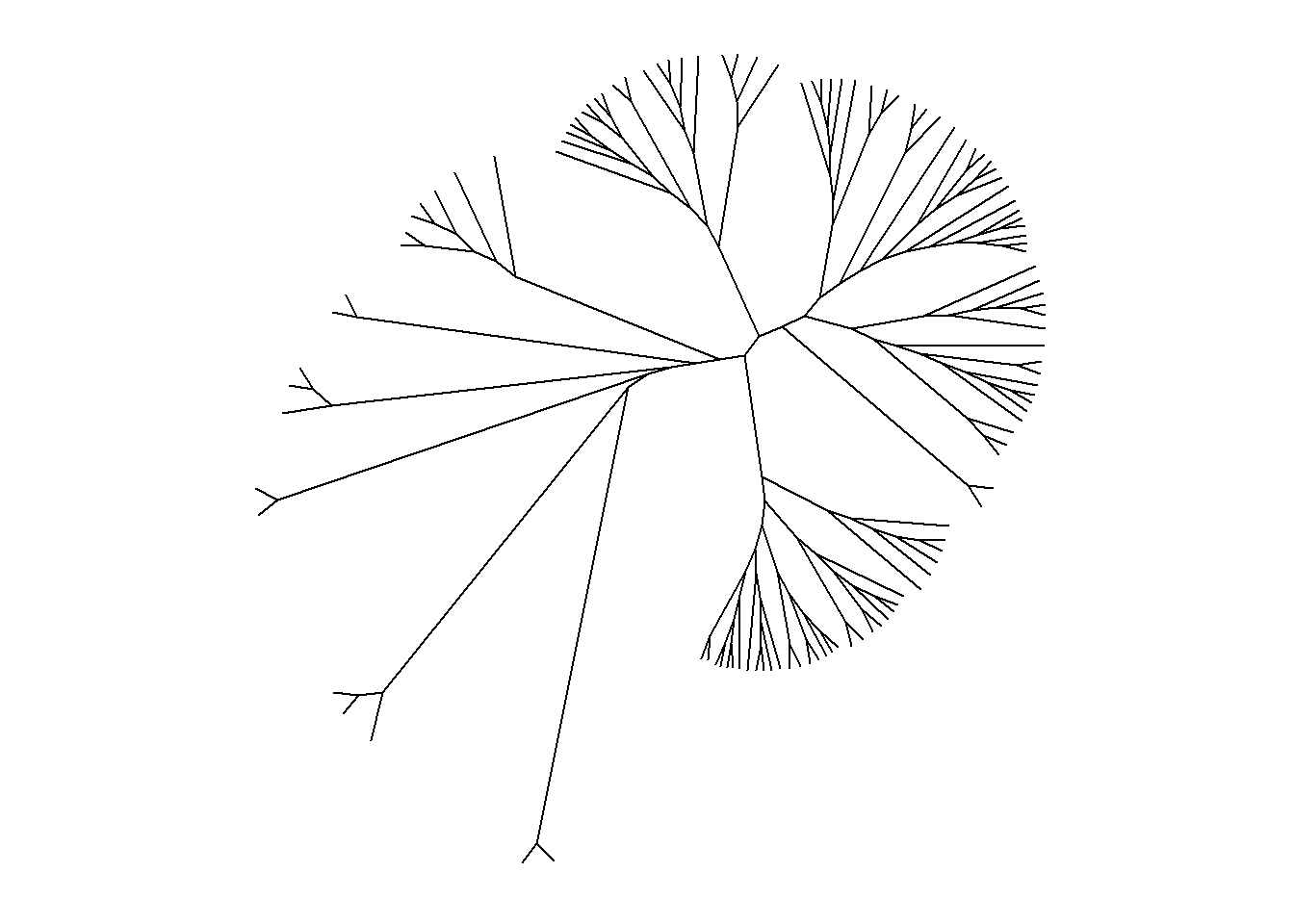
ggtree(arvore, layout = "daylight", branch.length = "none")
## Average angle change [1] 0.0616706965012774
## Average angle change [2] 0.0158681858153626
Cor, tipo de linha e espessura dos ramos
Podemos alterar o tipo de linha utilizado nos ramos (argumento linetype), cor dos ramos (color), e espessura dos ramos (size).
Vejamos:
ggtree(arvore, color = "blue", size = 1.5)
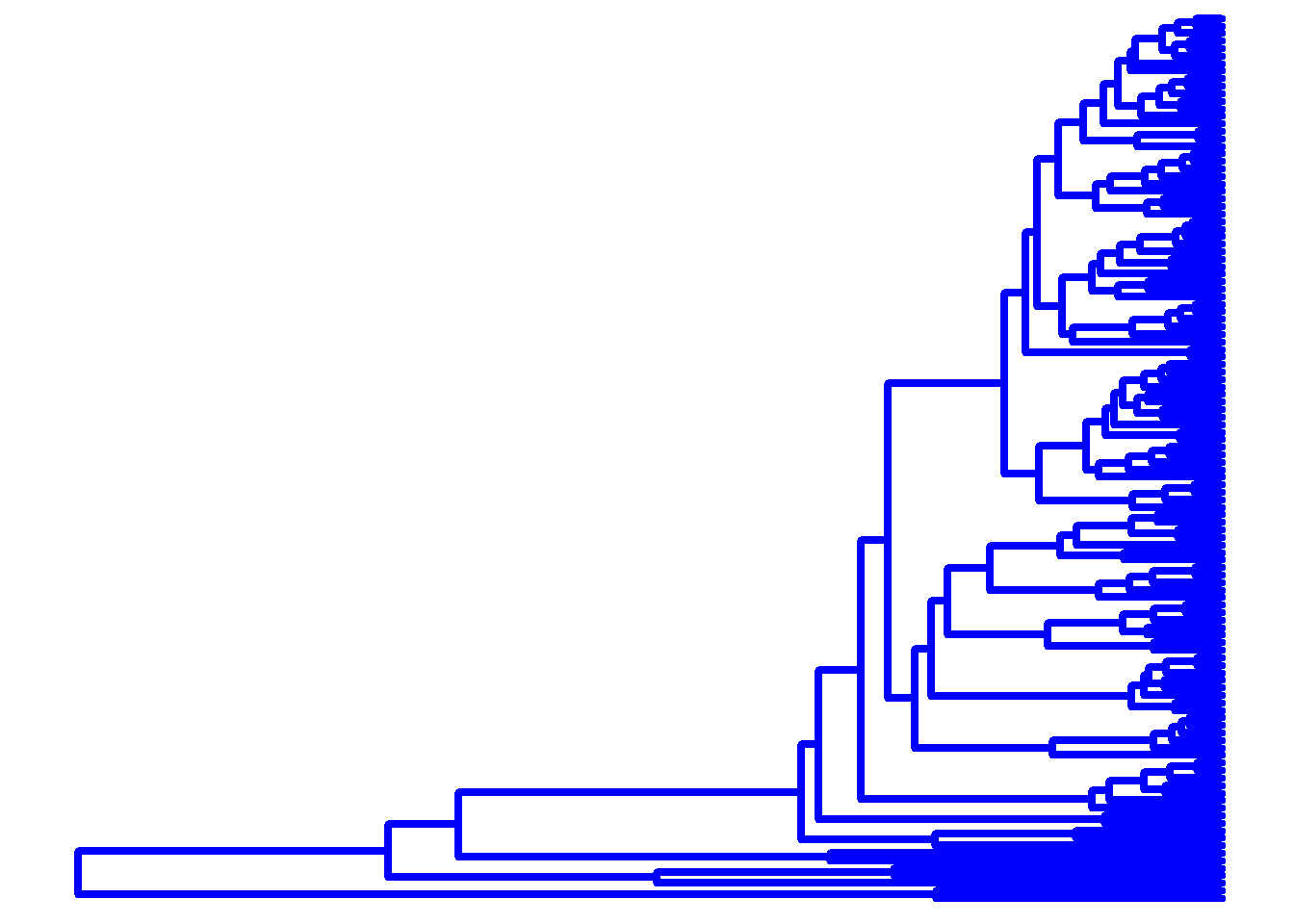
ggtree(arvore, linetype = "dotted", size = 2.2)

Inversão dos eixos X e Y
O pacote ggtree usa a mesma ‘gramática’ de visualização presente no pacote
ggplot2.
Portanto, utilizamos o operador + para adicionar as camadas de dados na árvore após o uso da função ggtree().
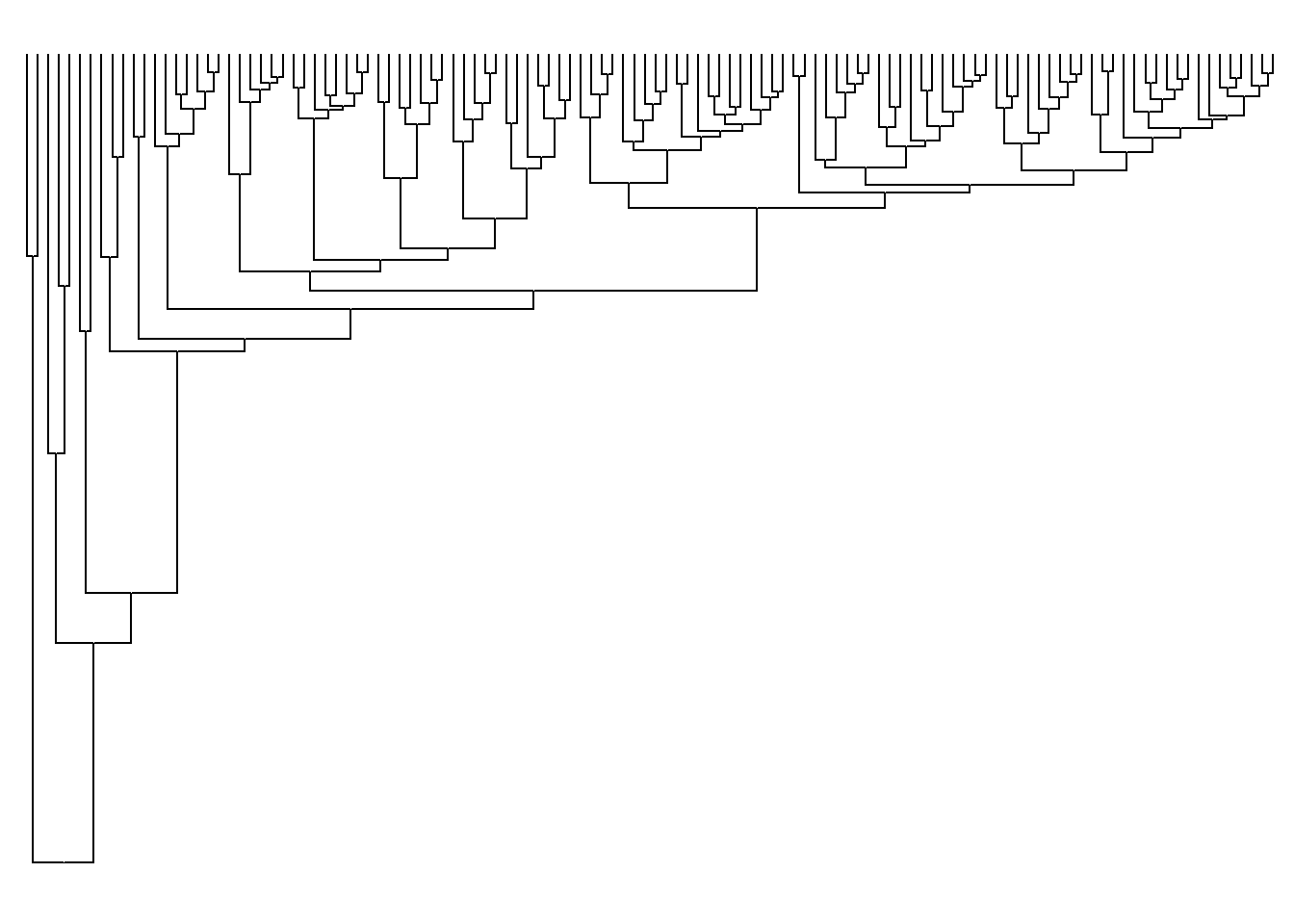
Podemos mudar o eixo de lugar.
No lugar de visualizarmos a árvore na horizontal, temos a árvore na vertical ao utilizar a função coord_flip().
ggtree(arvore) +
coord_flip()
Visualizando os nomes dos terminais
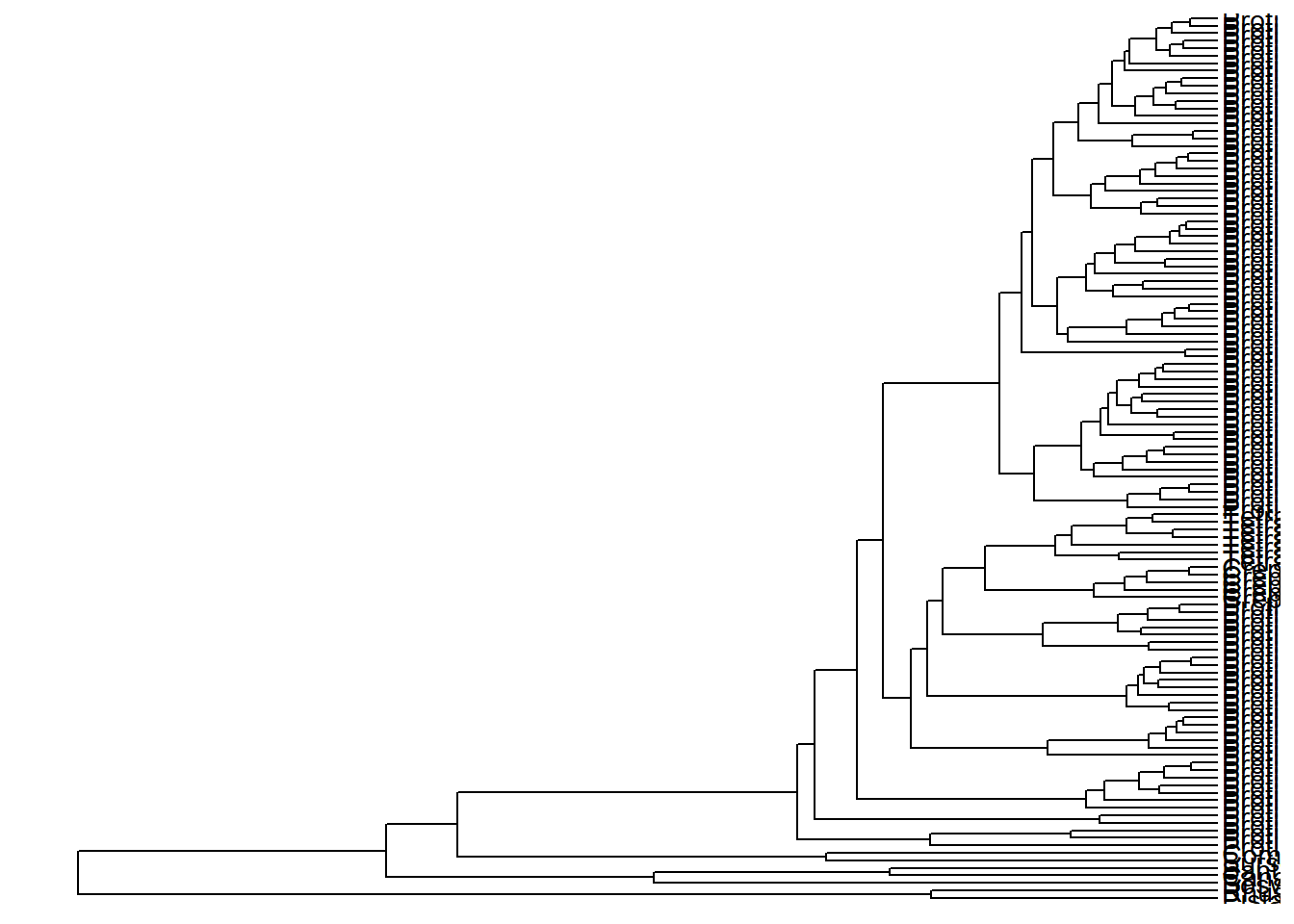
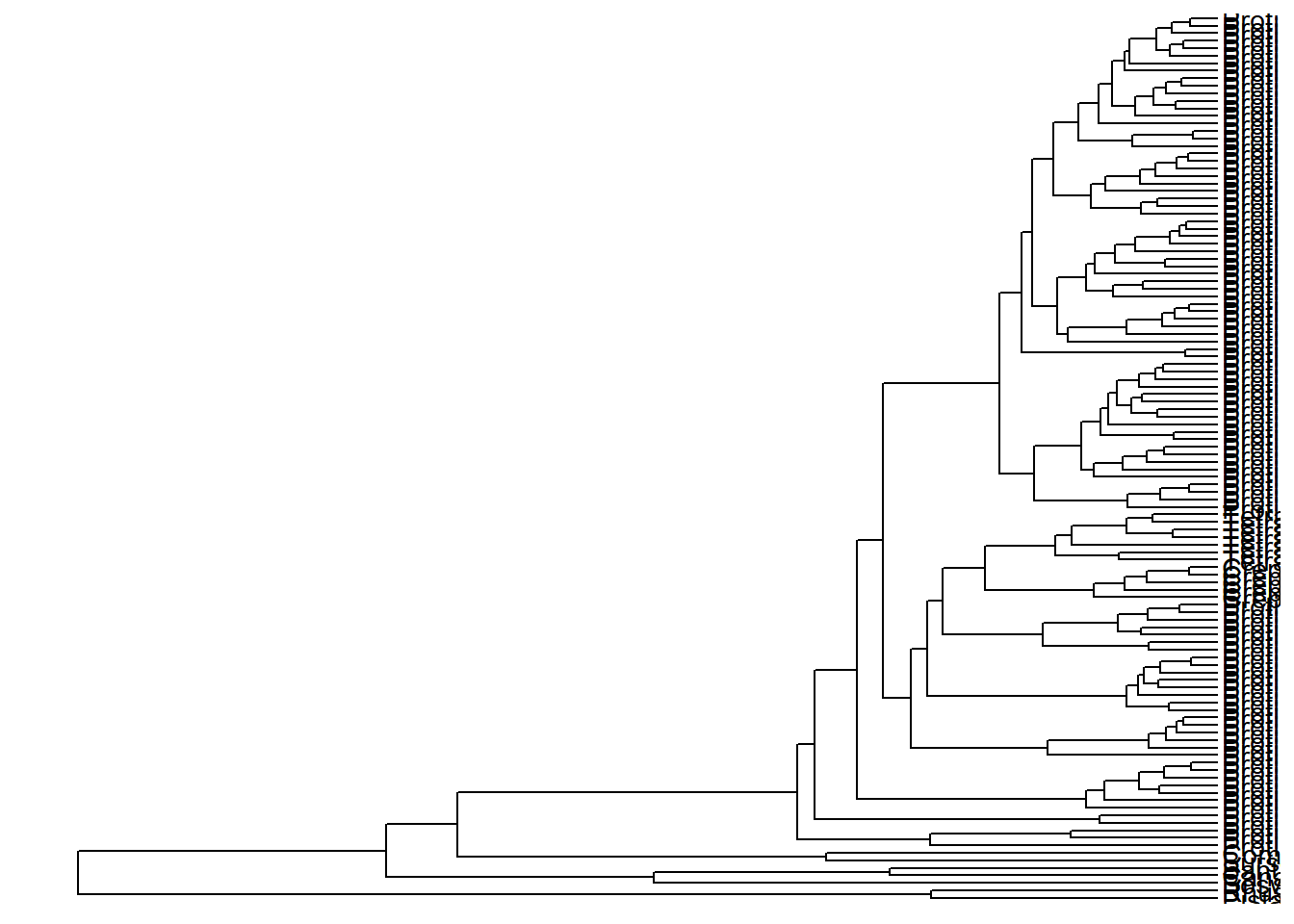
Usamos a função geom_tiplab() para visualizar o nome dos terminais:
ggtree(arvore) +
geom_tiplab()
Limites do eixo X para facilitar a visualização
Vejam que é difícil visualizar os nomes dos terminais pois tudo fica muito empacotado na imagem.
Há uma função para solucionar esta situação.
Ela se chama xlim_tree().
Com essa função, estabelecemos os limites do eixo X.
Executem os comandos abaixo e vejam a diferença entre os limites de X que coloquei.
Qual dos valores limites facilita a visualização? 30, 60, 90, ou 100?
ggtree(arvore) +
geom_tiplab() +
xlim_tree(c(0, 30))
ggtree(arvore) +
geom_tiplab() +
xlim_tree(c(0, 60))
ggtree(arvore) +
geom_tiplab() +
xlim_tree(c(0, 90))
ggtree(arvore) +
geom_tiplab() +
xlim_tree(c(0, 100))
Tamanho do nome dos terminais
Controlamos o tamanho da fonte do nome dos terminais com o argumento size da função geom_tiplab().
Vejamos:
ggtree(arvore) +
geom_tiplab(size = 1) +
xlim_tree(c(0, 100))

ggtree(arvore) +
geom_tiplab(size = 2) +
xlim_tree(c(0, 100))
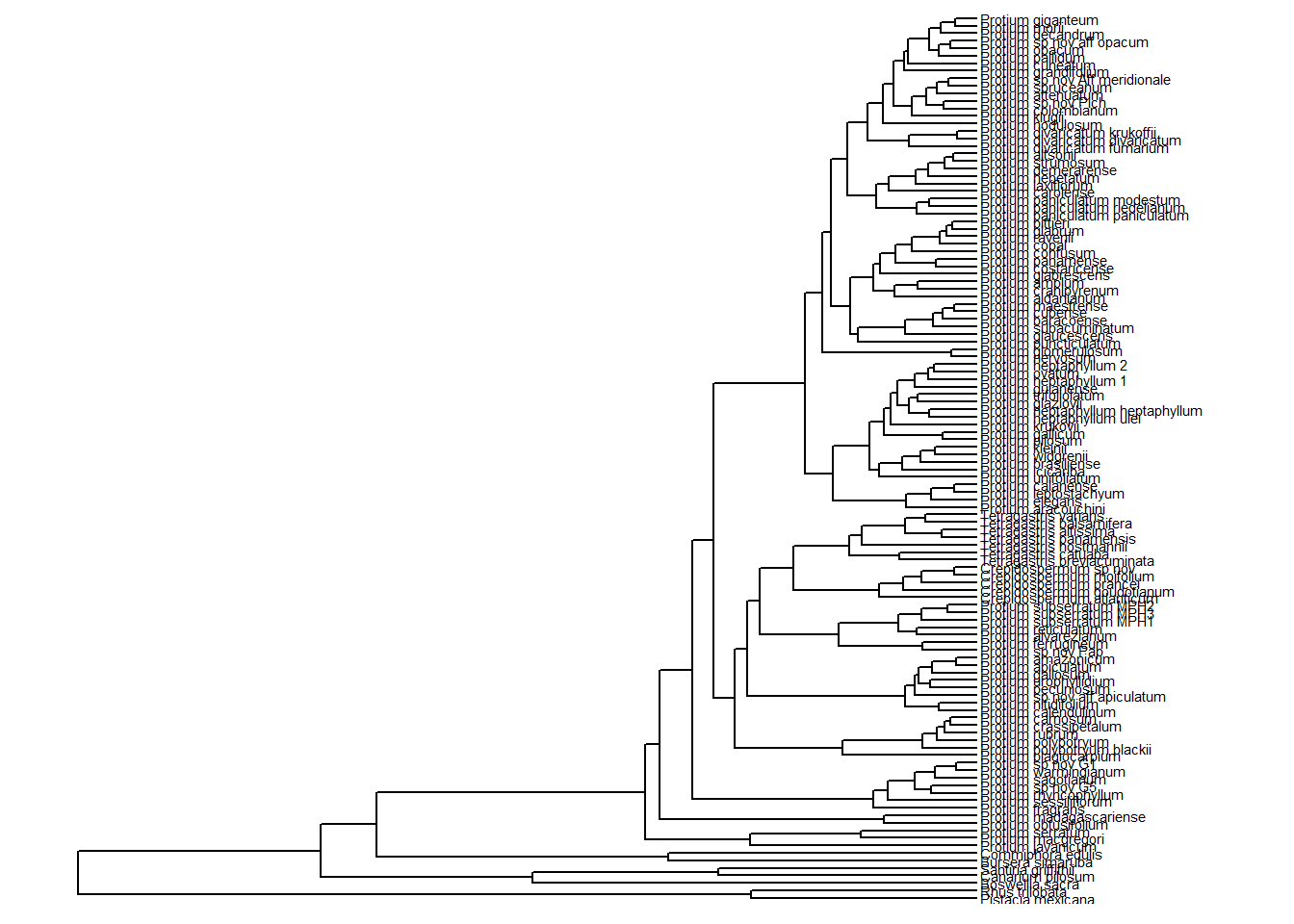
ggtree(arvore) +
geom_tiplab(size = 3) +
xlim_tree(c(0, 100))

Cores dos nomes dos terminais
Podemos também alterar a cor dos nomes dos terminais com o argumento color dentro da função geom_tiplab().
Vejamos:
ggtree(arvore) +
geom_tiplab(size = 2, color = "red") +
xlim_tree(c(0, 120))
ggtree(arvore) +
geom_tiplab(size = 3, color = "green") +
xlim_tree(c(0, 120))
ggtree(arvore) +
geom_tiplab(size = 4, color = "blue") +
xlim_tree(c(0, 120))
Mexendo com os nós
Para manipular os nós da árvore, usamos a função geom_nodepoint().
ggtree(arvore) +
geom_nodepoint()

Assim como a função geom_tiplab(), podemos mexer em cores (color), formas (shape) e intensidade da cor (alpha).
ggtree(arvore) +
geom_nodepoint(color="red", size = 2, shape = 17, alpha=.8)

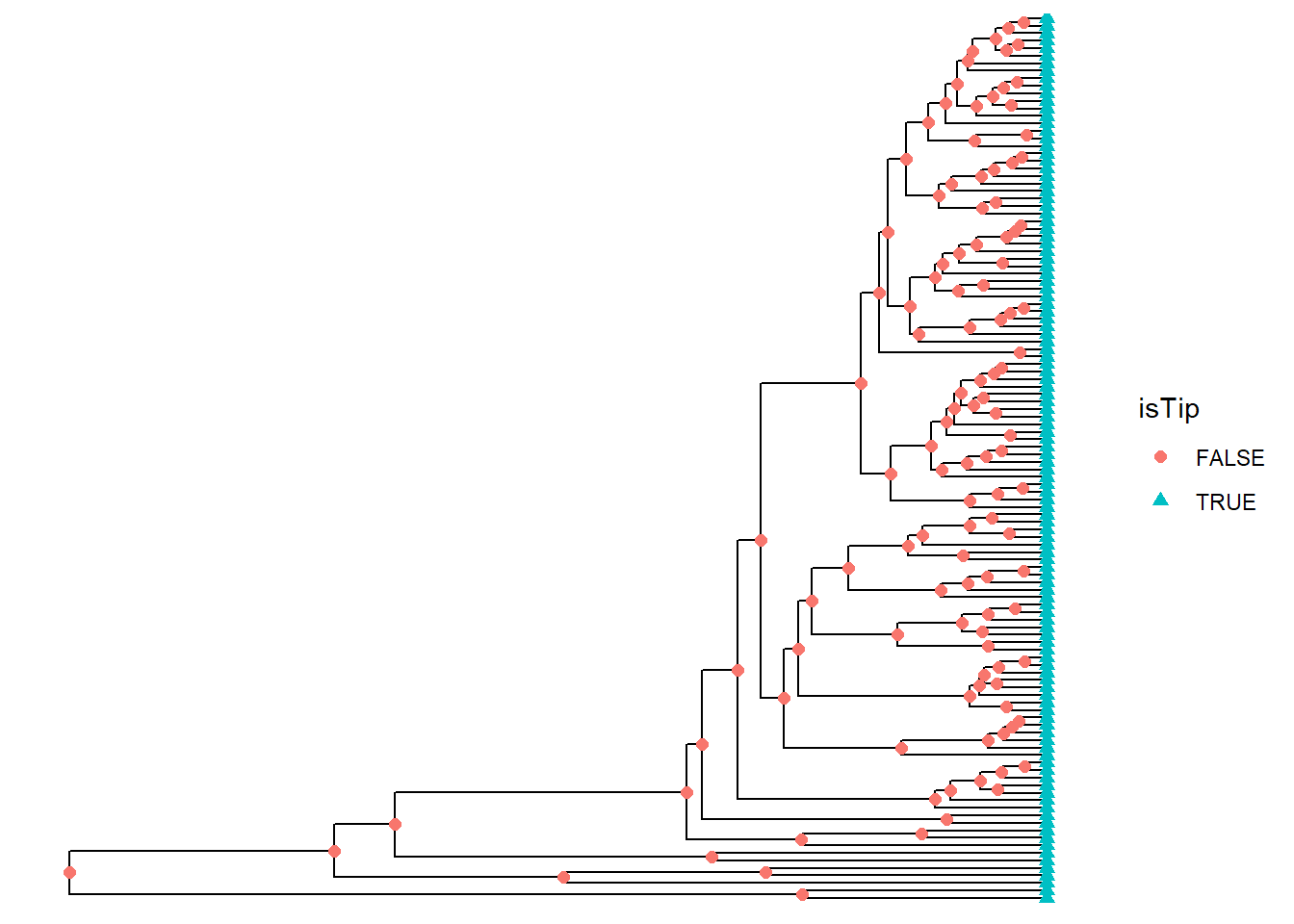
Forma dos nós e terminais
ggtree(arvore) +
geom_point(aes(shape = isTip, color = isTip), size = 2)
Filtrando a árvore por um determinado clado
Suponha que você quer visualizar apenas uma porção de sua árvore, um determinado clado de seu interesse, por exemplo.
O pacote tidytree (Yu 2023a) apresenta a função MRCA() que retorna o número do nó correspondente ao mais recente ancestral comum de um conjunto de espécies.
Desta forma, você é capaz de reduzir o seu universo amostral a um número reduzido de terminais.
Por exemplo, vamos buscar o ancestral comum mais recentes de Protium aracouchini (Aubl.) Marchand e de P. leptostachyum Cuatrec., um par de espécies que compõem o complexo P. aracouchini (Daly et al. 2022).
MRCA(p, .node1 = c("Protium aracouchini", "Protium leptostachyum"))
## [1] 206
A função retorna um número, que corresponde ao número do nó correspondente ao ancestral comum mais recente desse par de espécies.
Vamos salvar esse número em um objeto e fazer a redução de nossa árvore completa para apenas esse conjunto de espécies por meio da função tree_subset().
Vamos chamar essa árvore reduzida de arv_reduzida:
paracouch_no <- MRCA(p, .node1 = c("Protium aracouchini", "Protium leptostachyum"))
arv_reduzida <- tree_subset(arvore, node = paracouch_no, levels_back = 0)
arv_reduzida
## 'treedata' S4 object that stored information of
## 'S15484.nex'.
##
## ...@ phylo:
##
## Phylogenetic tree with 4 tips and 3 internal nodes.
##
## Tip labels:
## Protium elegans, Protium aracouchini, Protium calanense, Protium leptostachyum
##
## Rooted; includes branch lengths.
Agora vamos plotar esta árvore usando a função ggtree() e atribuí-la ao objeto cpa, abreviatura de complexo P. aracouchini:
cpa <- ggtree(arv_reduzida)
cpa
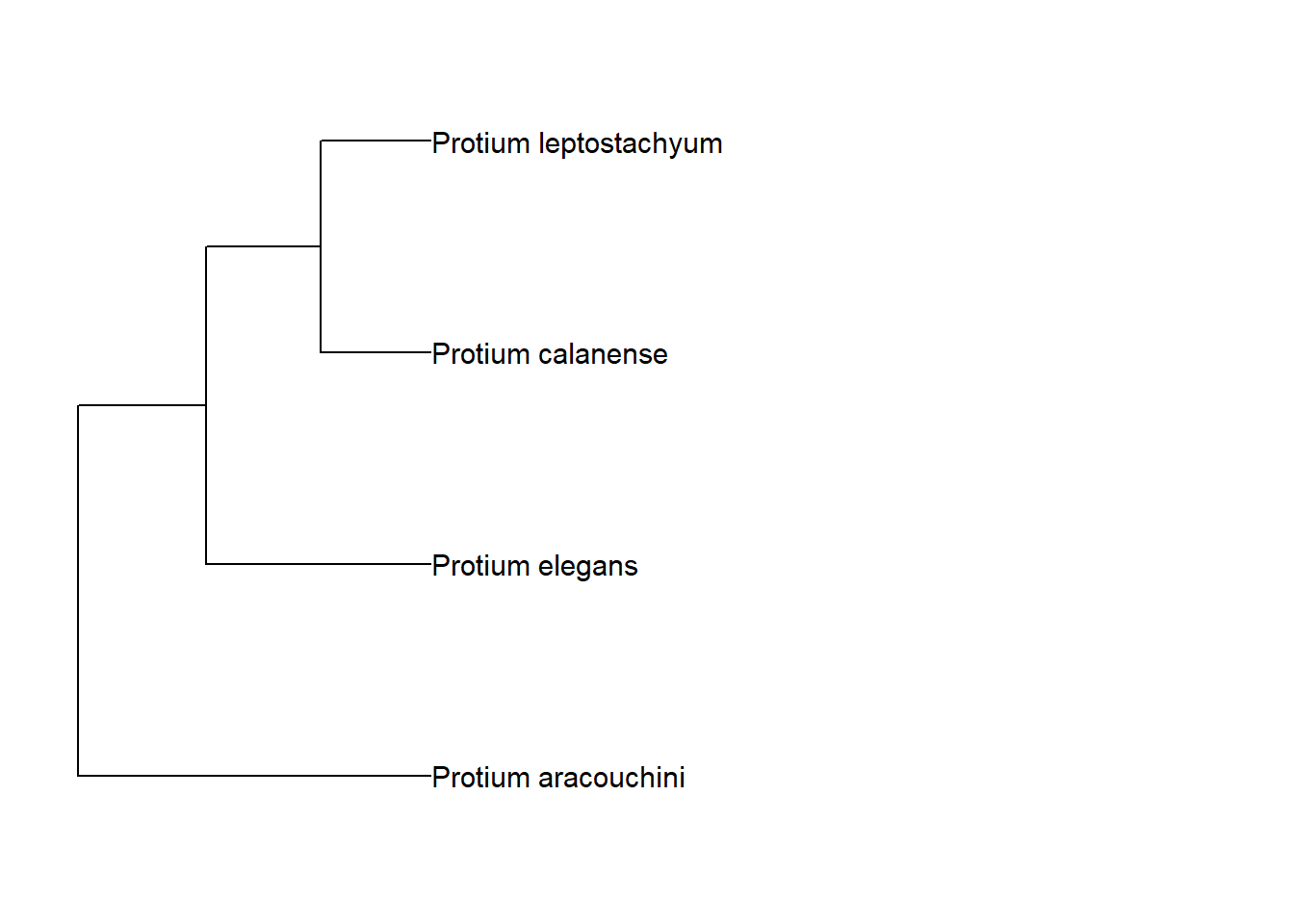
Lembrando o que aprendemos anteriormente, vamos plotar o nome dos terminais:
arv_cpa <- cpa +
geom_tiplab(size = 4) +
xlim_tree(c(0, 20))
arv_cpa
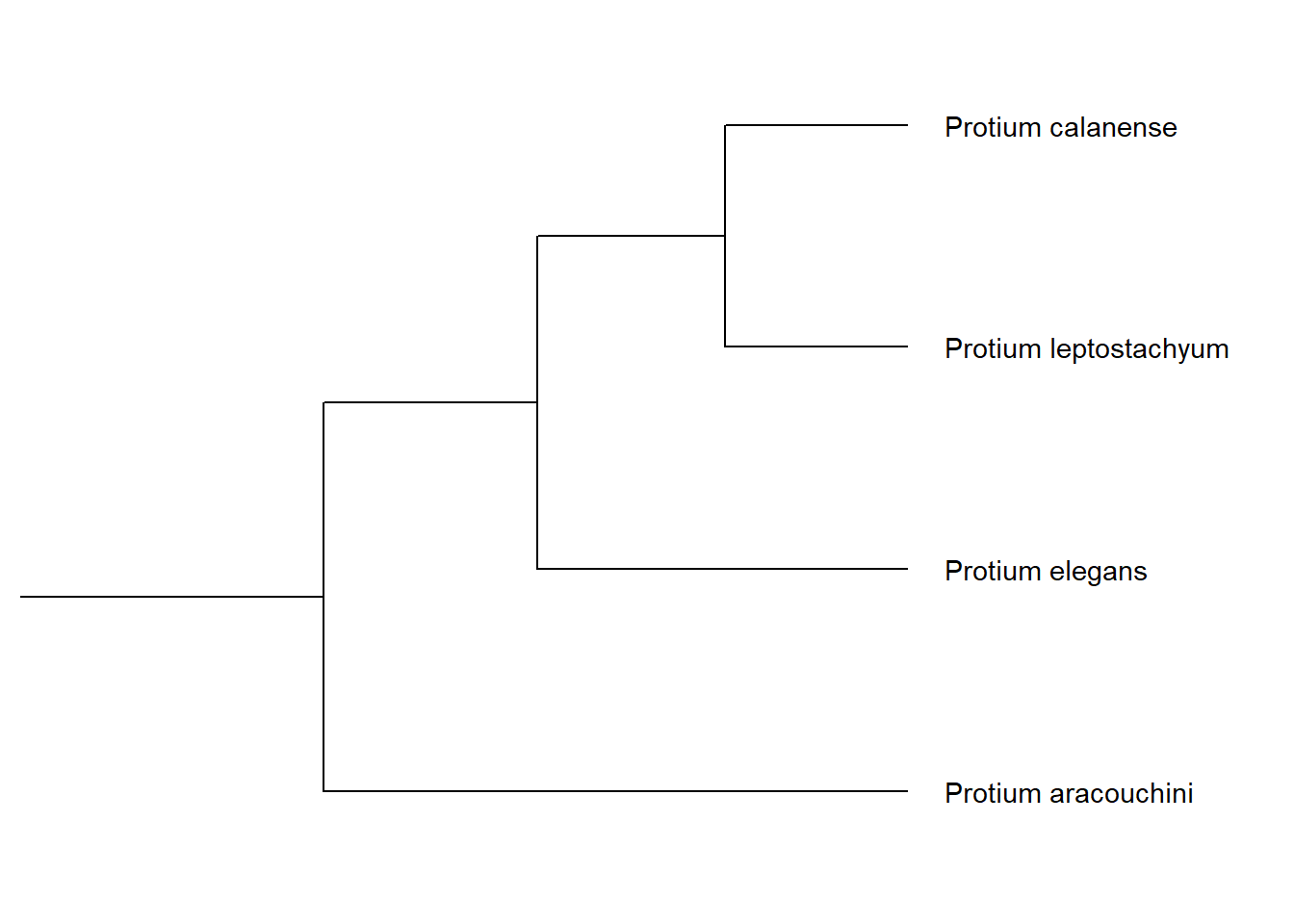
Visualização de um clado
Uma alternativa rápida à redução da árvore completa usando a função subset_tree() é escolher visualizar somente um determinado clado utilizando a função viewClade().
Vejamos:
paracouch_no <- MRCA(p, .node1 = c("Protium aracouchini", "Protium leptostachyum"))
viewClade(p, node = paracouch_no) + geom_tiplab()
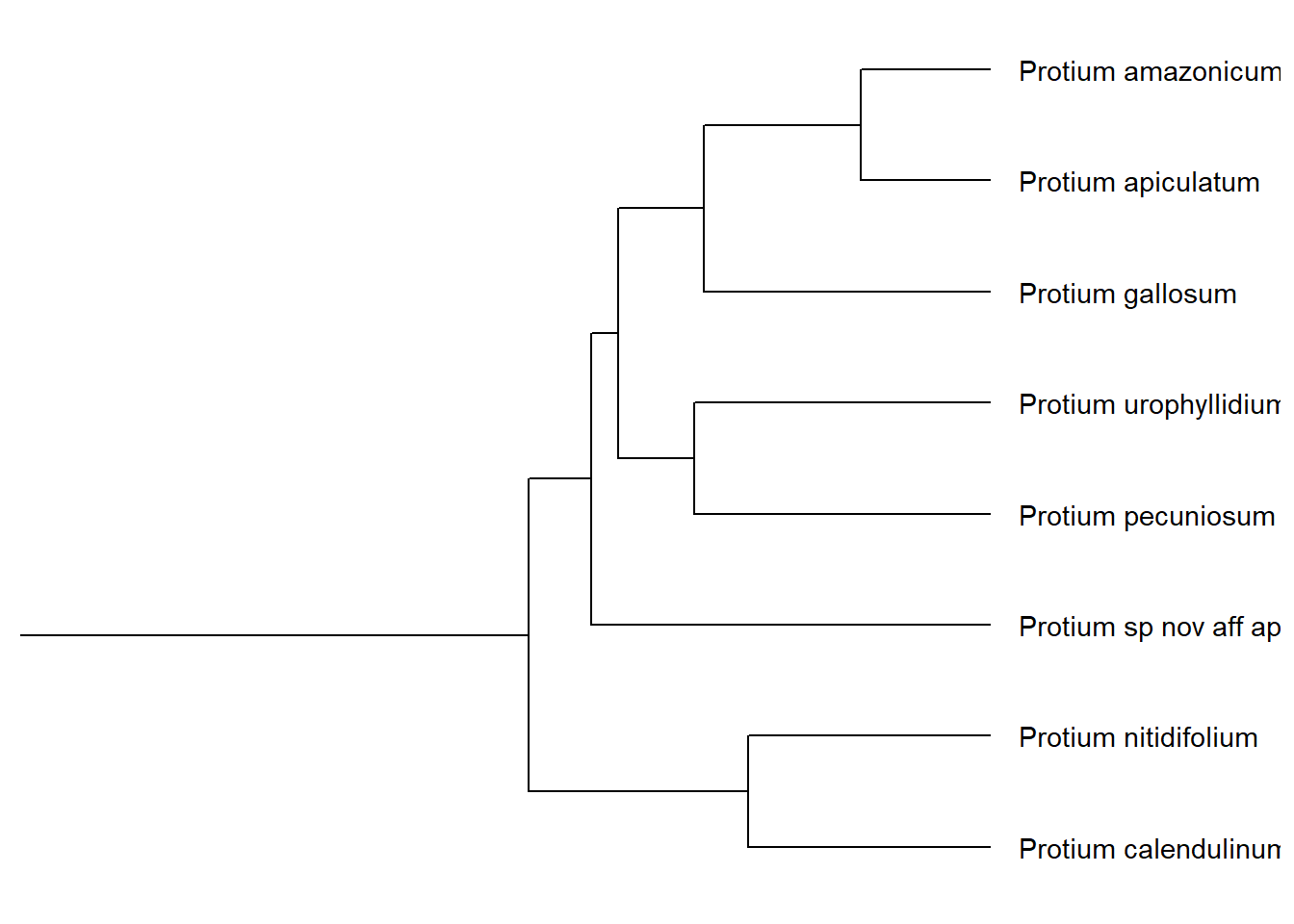
Agora, vamos ver outro clado.
Por exemplo, Protium seção Pepeanthos (Daly 2007).
Para isso, escolhi o nome de duas espécies do clado, P. calendulinum Daly e P. urophyllidium Daly e as inseri na função MRCA() utilizando nossa árvore p como modelo.
Usamos o resultado da função MRCA() no argumento node da função viewClade(), chegando assim ao clado desejado:
viewClade(p, node = MRCA(p, .node1 = c("Protium calendulinum", "Protium urophyllidium"))) +
geom_tiplab()
Mostrando a raiz
Você pode exibir a raiz de um clado usando a função geom_rootedge().
arv_cpa +
geom_rootedge()
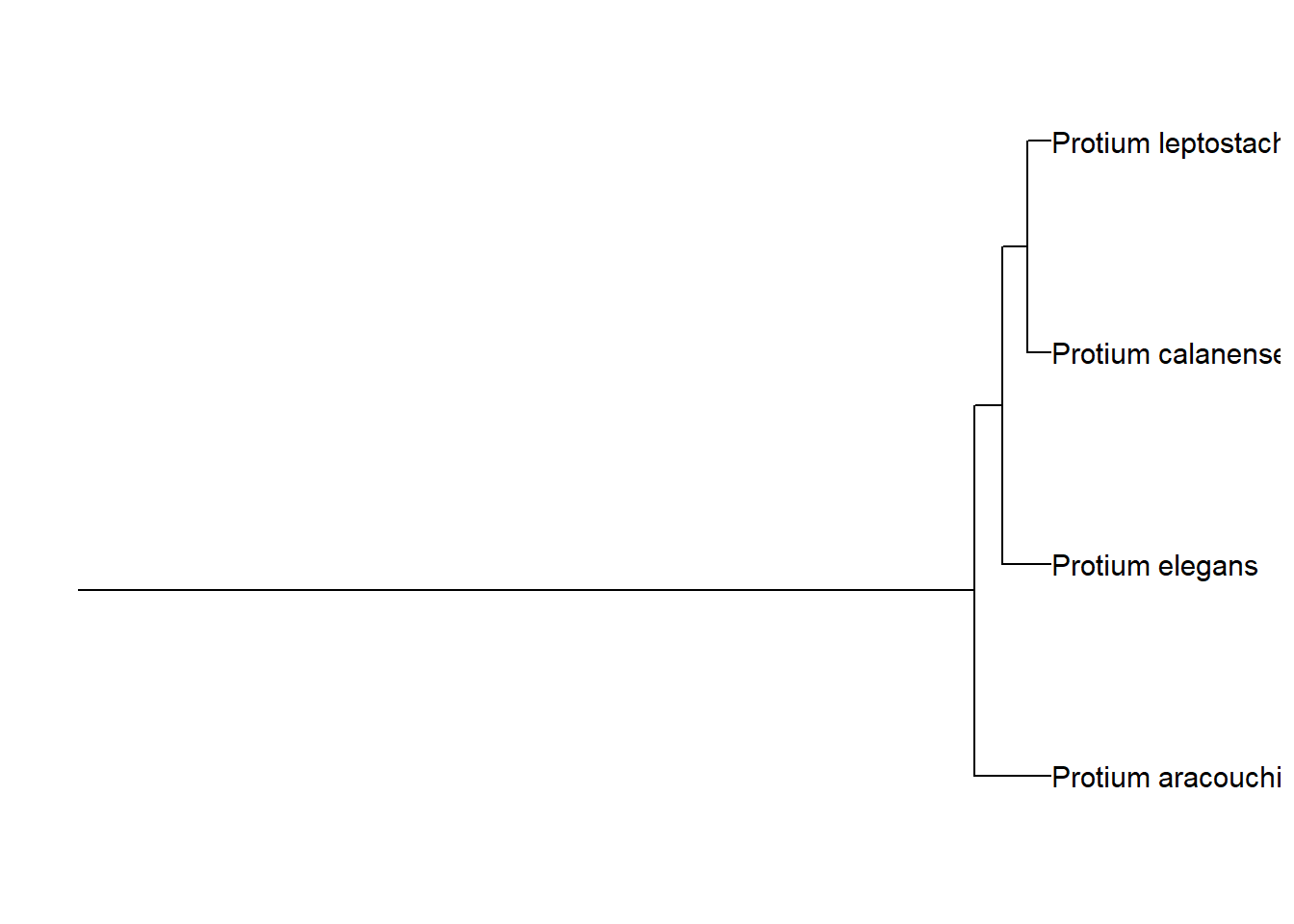
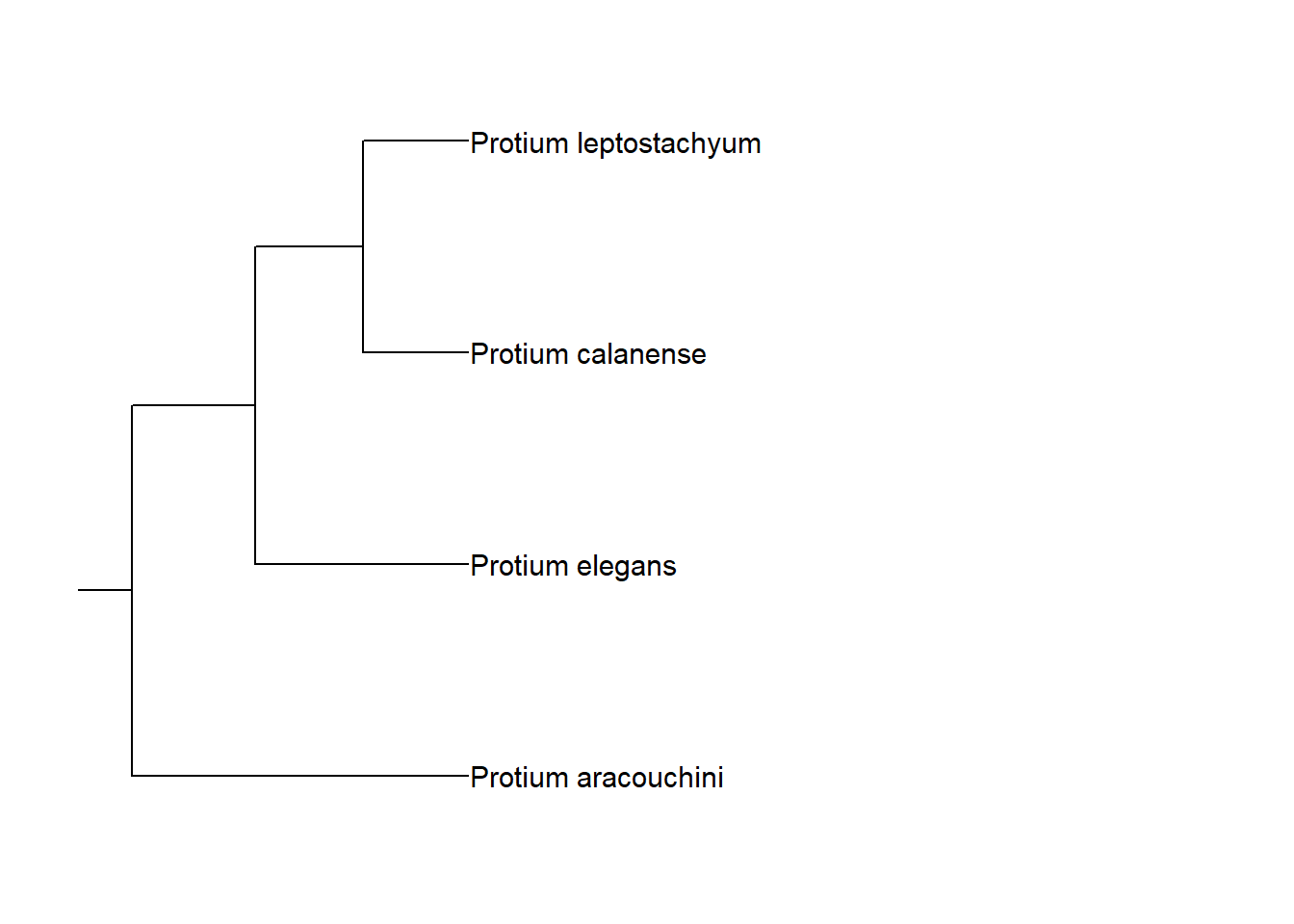
Você pode controlar o tamanho desse ramo por meio do argumento rootedge.
arv_cpa +
geom_rootedge(rootedge = 1)
Salvando a nossa árvore
Vamos agora utilizar o cowplot (Wilke 2020) para salvar nossa árvore. Aprendemos várias coisas, mas precisamos de algo mínimo para salvar. Que tal a árvore completa com os nomes dos terminais em um eixo X que nos permita enxergar corretamente os nomes das espécies? Façamos:
arvore_plot <-
ggtree(arvore) +
geom_tiplab(size = 1.2) +
xlim_tree(c(0, 100))
arvore_plot

Atenção deve ser dada ao tamanho da figura. É importante que você cheque as instruções de formatação de imagens para a revista a qual você pretende submeter seu artigo. Por exemplo, é comum que botânicos publiquem no periódico Brittonia, revista do Jardim Botânico de Nova Iorque. Esta revista aceita imagens ou com largura de 6.2 cm, que corresponde a uma coluna de uma página do periódico, ou largura de 13.8 cm, que corresponde à largura total da página. Já a altura não pode passar de 10 cm. Portanto, imaginando que submeteríamos essa imagem para essa revista, por se tratar de uma árvore filogenética, teríamos como largura 13.8 cm, e para manter uma proporção de 3:4, teríamos uma altura de 10.35 cm. Vamos escolher um dpi de 600 e salvar nossa imagem como um pdf. Basta então executar o comando abaixo:
cowplot::ggsave2(
plot = arvore_plot,
filename = "arvore_cheia.pdf",
dpi = 600,
width = 13.8,
height = 13.8*3/4,
units = "cm"
)
Compondo figuras
É comum quando lidamos com árvores filogenéticas apresentarmos uma árvore maior e um pedaço em destaque da árvore maior em algum lugar desta imagem.
O R permite que façamos isso na maior tranquilidade.
Por exemplo, temos a nossa árvore completa, arvore_plot, e a nossa árvore menor do complexo P. aracouchini, arv_cpa.
Vamos utilizar as funções ggdraw() e draw_plot() para combinar nossas imagens.
Na primeira função, ggdraw(), nós utilizamos nossa imagem maior, que servirá de pano de fundo para incluirmos a imagem menor em um pedaço da imagem da árvore completa.
Ao incluirmos a imagem menor, arv_cpa, por meio da função draw_plot(), devemos designar a posição dela no eixo x e y (valores de 0 a 1), além do tamanho da imagem em largura e altura (valores de 0 a 1).
Depois de inúmeras tentativas, cheguei a esses valores de x,y, largura e altura:
arvore_inset <- ggdraw(arvore_plot) +
draw_plot(arv_cpa,
x = .02,
y = .6,
width = .5,
height = .3)
arvore_inset

Quando vocês forem testar com os dados de vocês, vão tentando múltiplos valores até ver os que melhor ajustam sua imagem.
Para salvar a imagem, basta usar o comando abaixo, que um arquivo pdf de nome arvore_composta.pdf será salvo em sua pasta de trabalho.
cowplot::ggsave2(
plot = arvore_inset,
filename = "arvore_composta.pdf",
dpi = 600,
width = 13.8,
height = 13.8*3/4,
units = "cm"
)
Espero que tenham gostado. Deixem seus comentários, compartilhem, e vamos que vamos.
Referências
Daly, Douglas C. 2007. “A new section of Protium from the neotropics. Studies in neotropical Burseraceae XIII”. Brittonia 59 (1): 1–24. https://doi.org/10.1663/0007-196X(2007)59[1:ANSOPF]2.0.CO;2.
Daly, Douglas C., Ricardo Oliveira Perdiz, Paul Van Antwerp Fine, Gabriel Damasco, María Cristina Martínez-Habibe, e Laura Calvillo-Canadell. 2022. “A review of Neotropical Burseraceae”. Brazilian Journal of Botany, janeiro. https://doi.org/10.1007/s40415-021-00765-1.
Fine, Paul V. A., Felipe Zapata, e Douglas C. Daly. 2014. “Investigating processes of Neotropical rain forest tree diversification by examining the evolution and historical biogeography of the Protieae (Burseraceae)”. Evolution 68: 1988–2004. https://doi.org/10.1111/evo.12414.
Fine, Paul Van Antwerp, Felipe Zapata, e Douglas C. Daly. 2014. Dryad Dataset from “Investigating processes of neotropical rain forest tree diversification by examining the evolution and historical biogeography of the Protieae (Burseraceae)”. https://doi.org/10.5061/dryad.d71c1.
Morgan, Martin, e Marcel Ramos. 2023. BiocManager: Access the Bioconductor Project Package Repository. https://bioconductor.github.io/BiocManager/.
Paradis, Emmanuel, Simon Blomberg, Ben Bolker, Joseph Brown, Santiago Claramunt, Julien Claude, Hoa Sien Cuong, et al. 2023. ape: Analyses of Phylogenetics and Evolution. http://ape-package.ird.fr/.
Paradis, Emmanuel, e Klaus Schliep. 2019. “ape 5.0: an environment for modern phylogenetics and evolutionary analyses in R”. Bioinformatics 35: 526–28. https://doi.org/10.1093/bioinformatics/bty633.
Revell, Liam J. 2012. “phytools: An R package for phylogenetic comparative biology (and other things).” Methods in Ecology and Evolution 3: 217–23. https://doi.org/10.1111/j.2041-210X.2011.00169.x.
———. 2023. phytools: Phylogenetic Tools for Comparative Biology (and Other Things). https://github.com/liamrevell/phytools.
Wang, Li-Gen, Tommy Tsan-Yuk Lam, Shuangbin Xu, Zehan Dai, Lang Zhou, Tingze Feng, Pingfan Guo, et al. 2020. “treeio: an R package for phylogenetic tree input and output with richly annotated and associated data.” Molecular Biology and Evolution 37: 599–603. https://doi.org/10.1093/molbev/msz240.
Wickham, Hadley. 2016. ggplot2: Elegant Graphics for Data Analysis. Springer-Verlag New York. https://ggplot2.tidyverse.org.
Wickham, Hadley, Winston Chang, Lionel Henry, Thomas Lin Pedersen, Kohske Takahashi, Claus Wilke, Kara Woo, Hiroaki Yutani, e Dewey Dunnington. 2023. ggplot2: Create Elegant Data Visualisations Using the Grammar of Graphics. https://ggplot2.tidyverse.org.
Wilke, Claus O. 2020. cowplot: Streamlined Plot Theme and Plot Annotations for ggplot2. https://wilkelab.org/cowplot/.
Yu, Guangchuang. 2023a. tidytree: A Tidy Tool for Phylogenetic Tree Data Manipulation. https://www.amazon.com/Integration-Manipulation-Visualization-Phylogenetic-Computational-ebook/dp/B0B5NLZR1Z/.
———. 2023b. treeio: Base Classes and Functions for Phylogenetic Tree Input and Output. https://doi.org/10.18129/B9.bioc.treeio.
Yu, Guangchuang, Tommy Tsan-Yuk Lam, e Shuangbin Xu. 2023. ggtree: an R package for visualization of tree and annotation data. https://doi.org/10.18129/B9.bioc.ggtree.
Yu, Guangchuang, David Smith, Huachen Zhu, Yi Guan, e Tommy Tsan-Yuk Lam. 2017. “ggtree: an R package for visualization and annotation of phylogenetic trees with their covariates and other associated data.” Methods in Ecology and Evolution 8: 28–36. https://doi.org/10.1111/2041-210X.12628.